- Mô tả :
'ogbg-molpcba' là bộ dữ liệu phân tử được lấy mẫu từ PubChem BioAssay. Nó là một bộ dữ liệu dự đoán đồ thị từ Open Graph Benchmark (OGB).
Tập dữ liệu này là thử nghiệm và API có thể thay đổi trong các bản phát hành trong tương lai.
Mô tả dưới đây của bộ dữ liệu được điều chỉnh từ bài báo OGB:
Định dạng đầu vào
Tất cả các phân tử được xử lý trước bằng RDKit ([1]).
- Mỗi biểu đồ đại diện cho một phân tử, trong đó các nút là nguyên tử và các cạnh là liên kết hóa học.
- Các tính năng nút đầu vào là 9 chiều, chứa số nguyên tử và tính đối xứng, cũng như các tính năng nguyên tử bổ sung khác như điện tích chính thức và liệu nguyên tử có nằm trong vòng hay không.
- Các tính năng cạnh đầu vào là 3 chiều, chứa loại liên kết, hóa học lập thể của liên kết, cũng như một tính năng liên kết bổ sung cho biết liệu liên kết có được liên hợp hay không.
Mô tả chính xác của tất cả các tính năng có tại https://github.com/snap-stanford/ogb/blob/master/ogb/utils/features.py
Sự dự đoán
Nhiệm vụ là dự đoán 128 hoạt động sinh học khác nhau (không hoạt động/hoạt động). Xem [2] và [3] để biết thêm mô tả về các mục tiêu này. Không phải tất cả các mục tiêu đều áp dụng cho từng phân tử: các mục tiêu bị thiếu được chỉ định bởi NaN.
Người giới thiệu
[1]: Greg Landrum, et al. 'RDKit: Hóa tin học mã nguồn mở'. URL: https://github.com/rdkit/rdkit
[2]: Bharath Ramsundar, Steven Kearnes, Patrick Riley, Dale Webster, David Konerding và Vijay Pande. 'Mạng đa nhiệm lớn để khám phá thuốc'. URL: https://arxiv.org/pdf/1502.02072.pdf
[3]: Zhenqin Wu, Bharath Ramsundar, Evan N Feinberg, Joseph Gomes, Caleb Geniesse, Aneesh S. Pappu, Karl Leswing, và Vijay Pande. MoleculeNet: điểm chuẩn cho học máy phân tử. Khoa học Hóa học, 9(2):513-530, 2018.
Trang chủ : https://ogb.stanford.edu/docs/graphprop
Mã nguồn :
tfds.datasets.ogbg_molpcba.BuilderPhiên bản :
-
0.1.0: Phiên bản đầu tiên của API thử nghiệm. -
0.1.1: Hiển thị rõ ràng số cạnh trong mỗi biểu đồ. -
0.1.2: Thêm trường siêu dữ liệu cho GraphVisualizer. -
0.1.3(mặc định): Thêm trường siêu dữ liệu cho tên của các tác vụ riêng lẻ.
-
Kích thước tải xuống :
37.70 MiBKích thước tập dữ liệu :
822.53 MiBTự động lưu vào bộ nhớ cache ( tài liệu ): Không
Chia tách :
| Tách ra | ví dụ |
|---|---|
'test' | 43,793 |
'train' | 350,343 |
'validation' | 43,793 |
- Cấu trúc tính năng :
FeaturesDict({
'edge_feat': Tensor(shape=(None, 3), dtype=float32),
'edge_index': Tensor(shape=(None, 2), dtype=int64),
'labels': Tensor(shape=(128,), dtype=float32),
'node_feat': Tensor(shape=(None, 9), dtype=float32),
'num_edges': Tensor(shape=(None,), dtype=int64),
'num_nodes': Tensor(shape=(None,), dtype=int64),
})
- Tài liệu tính năng :
| Tính năng | Lớp học | Hình dạng | Dtype | Sự miêu tả |
|---|---|---|---|---|
| Tính năngDict | ||||
| cạnh_feat | tenxơ | (Không, 3) | phao32 | |
| cạnh_index | tenxơ | (Không, 2) | int64 | |
| nhãn | tenxơ | (128,) | phao32 | |
| Không thất bại | tenxơ | (Không, 9) | phao32 | |
| số_cạnh | tenxơ | (Không có,) | int64 | |
| num_nodes | tenxơ | (Không có,) | int64 |
Các khóa được giám sát (Xem
as_superviseddoc ):NoneHình ( tfds.show_examples ):
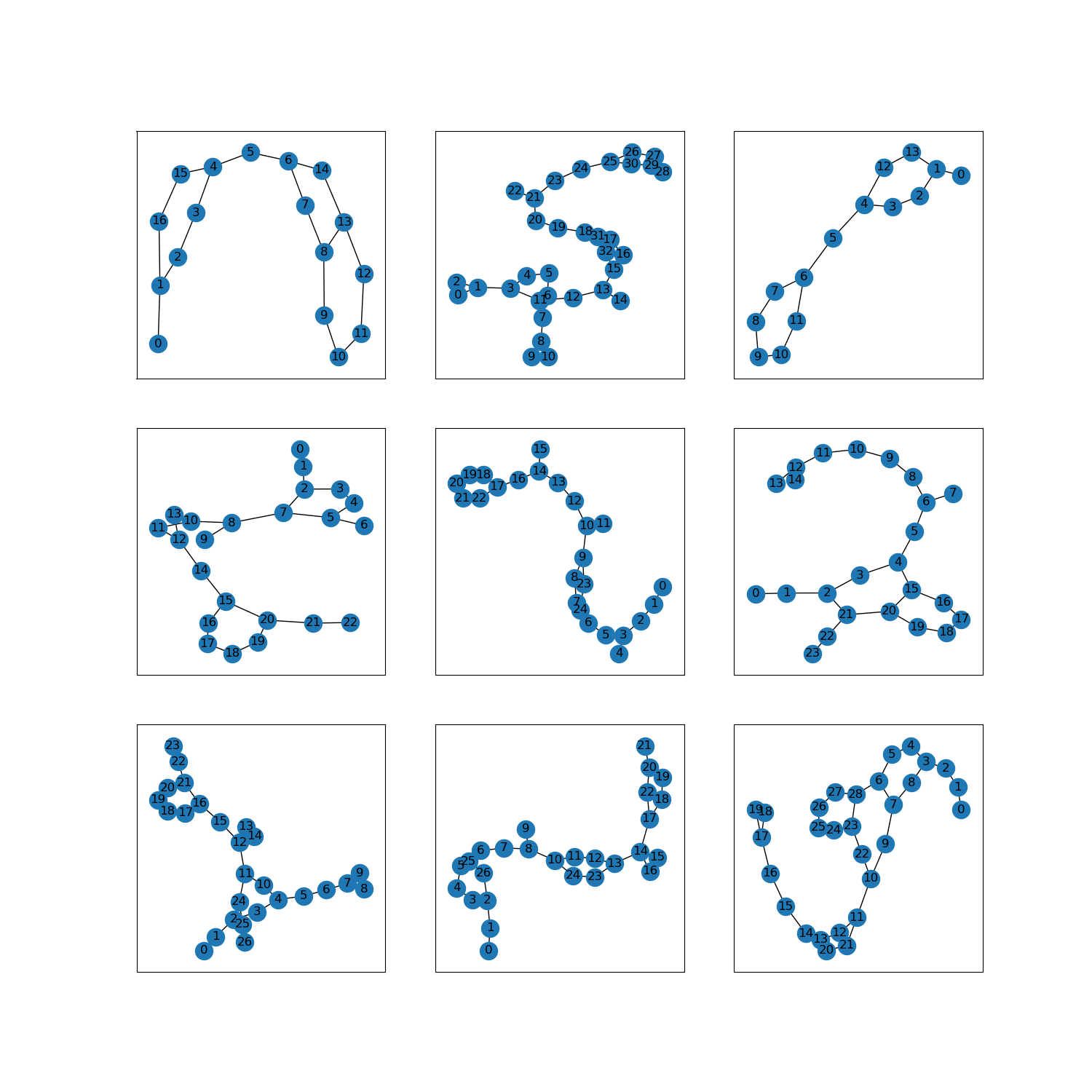
- Ví dụ ( tfds.as_dataframe ):
- trích dẫn :
@inproceedings{DBLP:conf/nips/HuFZDRLCL20,
author = {Weihua Hu and
Matthias Fey and
Marinka Zitnik and
Yuxiao Dong and
Hongyu Ren and
Bowen Liu and
Michele Catasta and
Jure Leskovec},
editor = {Hugo Larochelle and
Marc Aurelio Ranzato and
Raia Hadsell and
Maria{-}Florina Balcan and
Hsuan{-}Tien Lin},
title = {Open Graph Benchmark: Datasets for Machine Learning on Graphs},
booktitle = {Advances in Neural Information Processing Systems 33: Annual Conference
on Neural Information Processing Systems 2020, NeurIPS 2020, December
6-12, 2020, virtual},
year = {2020},
url = {https://proceedings.neurips.cc/paper/2020/hash/fb60d411a5c5b72b2e7d3527cfc84fd0-Abstract.html},
timestamp = {Tue, 19 Jan 2021 15:57:06 +0100},
biburl = {https://dblp.org/rec/conf/nips/HuFZDRLCL20.bib},
bibsource = {dblp computer science bibliography, https://dblp.org}
}

