 Ver en TensorFlow.org Ver en TensorFlow.org |  Ejecutar en Google Colab Ejecutar en Google Colab |  Ver fuente en GitHub Ver fuente en GitHub |  Descargar cuaderno Descargar cuaderno |
Análisis de componentes principales probabilístico (PCA) es una técnica de reducción de la dimensionalidad que analiza los datos a través de un espacio latente inferior dimensional ( Tipping y Bishop 1999 ). Se utiliza a menudo cuando faltan valores en los datos o para escalado multidimensional.
Importaciones
import functools
import warnings
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
import tensorflow.compat.v2 as tf
import tensorflow_probability as tfp
from tensorflow_probability import bijectors as tfb
from tensorflow_probability import distributions as tfd
tf.enable_v2_behavior()
plt.style.use("ggplot")
warnings.filterwarnings('ignore')
El modelo
Considere un conjunto de datos \(\mathbf{X} = \{\mathbf{x}_n\}\) de \(N\) puntos de datos, donde cada punto de datos es \(D\)dimensional, $ \ vec {x} _n \ in \ mathbb {R} ^ D\(. We aim to represent each \)\ vec {x} _n $ bajo una variable latente \(\mathbf{z}_n \in \mathbb{R}^K\) con dimensión inferior, $ K <D\(. The set of principal axes \)\ mathbf {W} $ relaciona las variables latentes a los datos.
Específicamente, asumimos que cada variable latente se distribuye normalmente,
\[ \begin{equation*} \mathbf{z}_n \sim N(\mathbf{0}, \mathbf{I}). \end{equation*} \]
El punto de datos correspondiente se genera a través de una proyección,
\[ \begin{equation*} \mathbf{x}_n \mid \mathbf{z}_n \sim N(\mathbf{W}\mathbf{z}_n, \sigma^2\mathbf{I}), \end{equation*} \]
donde la matriz \(\mathbf{W}\in\mathbb{R}^{D\times K}\) se conoce como los ejes principales. En PCA probabilística, estamos interesados normalmente en la estimación de los ejes principales \(\mathbf{W}\) y el término de ruido\(\sigma^2\).
El PCA probabilístico generaliza el PCA clásico. Al marginar la variable latente, la distribución de cada punto de datos es
\[ \begin{equation*} \mathbf{x}_n \sim N(\mathbf{0}, \mathbf{W}\mathbf{W}^\top + \sigma^2\mathbf{I}). \end{equation*} \]
PCA clásico es el caso específico de PCA probabilístico cuando la covarianza del ruido se hace, infinitesimalmente pequeña \(\sigma^2 \to 0\).
Configuramos nuestro modelo a continuación. En nuestro análisis, suponemos \(\sigma\) es conocido, y en lugar de punto estimar \(\mathbf{W}\) como un parámetro de modelo, colocamos un previo sobre él con el fin de inferir una distribución más ejes principales. Vamos a expresar el modelo como la PTF JointDistribution, en concreto, vamos a utilizar JointDistributionCoroutineAutoBatched .
def probabilistic_pca(data_dim, latent_dim, num_datapoints, stddv_datapoints):
w = yield tfd.Normal(loc=tf.zeros([data_dim, latent_dim]),
scale=2.0 * tf.ones([data_dim, latent_dim]),
name="w")
z = yield tfd.Normal(loc=tf.zeros([latent_dim, num_datapoints]),
scale=tf.ones([latent_dim, num_datapoints]),
name="z")
x = yield tfd.Normal(loc=tf.matmul(w, z),
scale=stddv_datapoints,
name="x")
num_datapoints = 5000
data_dim = 2
latent_dim = 1
stddv_datapoints = 0.5
concrete_ppca_model = functools.partial(probabilistic_pca,
data_dim=data_dim,
latent_dim=latent_dim,
num_datapoints=num_datapoints,
stddv_datapoints=stddv_datapoints)
model = tfd.JointDistributionCoroutineAutoBatched(concrete_ppca_model)
Los datos
Podemos utilizar el modelo para generar datos mediante el muestreo de la distribución previa conjunta.
actual_w, actual_z, x_train = model.sample()
print("Principal axes:")
print(actual_w)
Principal axes: tf.Tensor( [[ 2.2801023] [-1.1619819]], shape=(2, 1), dtype=float32)
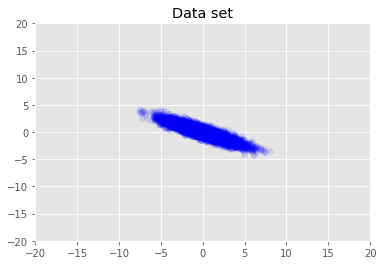
Visualizamos el conjunto de datos.
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1)
plt.axis([-20, 20, -20, 20])
plt.title("Data set")
plt.show()
Inferencia máxima a posteriori
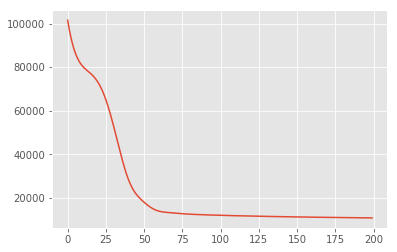
Primero buscamos la estimación puntual de las variables latentes que maximiza la densidad de probabilidad posterior. Esto se conoce como máximo una inferencia (MAP) posteriori, y se realiza mediante el cálculo de los valores de \(\mathbf{W}\) y \(\mathbf{Z}\) que maximizan la densidad posterior \(p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X}) \propto p(\mathbf{W}, \mathbf{Z}, \mathbf{X})\).
w = tf.Variable(tf.random.normal([data_dim, latent_dim]))
z = tf.Variable(tf.random.normal([latent_dim, num_datapoints]))
target_log_prob_fn = lambda w, z: model.log_prob((w, z, x_train))
losses = tfp.math.minimize(
lambda: -target_log_prob_fn(w, z),
optimizer=tf.optimizers.Adam(learning_rate=0.05),
num_steps=200)
plt.plot(losses)
[<matplotlib.lines.Line2D at 0x7f19897a42e8>]
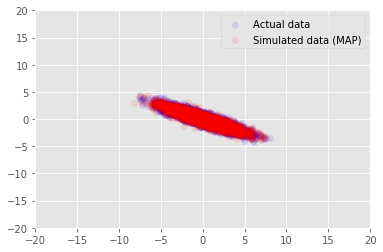
Podemos utilizar el modelo de datos de ejemplo para los valores inferidos para \(\mathbf{W}\) y \(\mathbf{Z}\), y compare con el conjunto de datos real que condicionado a.
print("MAP-estimated axes:")
print(w)
_, _, x_generated = model.sample(value=(w, z, None))
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1, label='Actual data')
plt.scatter(x_generated[0, :], x_generated[1, :], color='red', alpha=0.1, label='Simulated data (MAP)')
plt.legend()
plt.axis([-20, 20, -20, 20])
plt.show()
MAP-estimated axes:
<tf.Variable 'Variable:0' shape=(2, 1) dtype=float32, numpy=
array([[ 2.9135954],
[-1.4826864]], dtype=float32)>
Inferencia variacional
MAP se puede utilizar para encontrar el modo (o uno de los modos) de la distribución posterior, pero no proporciona ninguna otra información al respecto. A continuación utilizamos la inferencia variacional, donde el Distribtion posterior \(p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X})\) se aproxima mediante una distribución variacional \(q(\mathbf{W}, \mathbf{Z})\) parametrizado por \(\boldsymbol{\lambda}\). El objetivo es encontrar los parámetros variacionales \(\boldsymbol{\lambda}\) que reducen al mínimo la divergencia KL entre Q y la parte posterior, \(\mathrm{KL}(q(\mathbf{W}, \mathbf{Z}) \mid\mid p(\mathbf{W}, \mathbf{Z} \mid \mathbf{X}))\), o equivalentemente, que maximizan la evidencia límite inferior, \(\mathbb{E}_{q(\mathbf{W},\mathbf{Z};\boldsymbol{\lambda})}\left[ \log p(\mathbf{W},\mathbf{Z},\mathbf{X}) - \log q(\mathbf{W},\mathbf{Z}; \boldsymbol{\lambda}) \right]\).
qw_mean = tf.Variable(tf.random.normal([data_dim, latent_dim]))
qz_mean = tf.Variable(tf.random.normal([latent_dim, num_datapoints]))
qw_stddv = tfp.util.TransformedVariable(1e-4 * tf.ones([data_dim, latent_dim]),
bijector=tfb.Softplus())
qz_stddv = tfp.util.TransformedVariable(
1e-4 * tf.ones([latent_dim, num_datapoints]),
bijector=tfb.Softplus())
def factored_normal_variational_model():
qw = yield tfd.Normal(loc=qw_mean, scale=qw_stddv, name="qw")
qz = yield tfd.Normal(loc=qz_mean, scale=qz_stddv, name="qz")
surrogate_posterior = tfd.JointDistributionCoroutineAutoBatched(
factored_normal_variational_model)
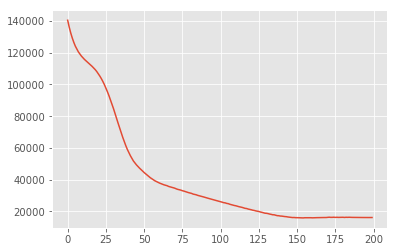
losses = tfp.vi.fit_surrogate_posterior(
target_log_prob_fn,
surrogate_posterior=surrogate_posterior,
optimizer=tf.optimizers.Adam(learning_rate=0.05),
num_steps=200)
print("Inferred axes:")
print(qw_mean)
print("Standard Deviation:")
print(qw_stddv)
plt.plot(losses)
plt.show()
Inferred axes:
<tf.Variable 'Variable:0' shape=(2, 1) dtype=float32, numpy=
array([[ 2.4168603],
[-1.2236133]], dtype=float32)>
Standard Deviation:
<TransformedVariable: dtype=float32, shape=[2, 1], fn="softplus", numpy=
array([[0.0042499 ],
[0.00598824]], dtype=float32)>
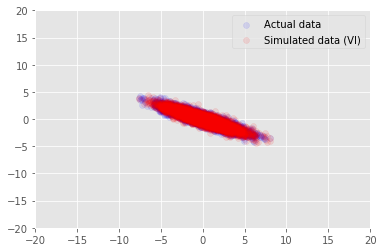
posterior_samples = surrogate_posterior.sample(50)
_, _, x_generated = model.sample(value=(posterior_samples))
# It's a pain to plot all 5000 points for each of our 50 posterior samples, so
# let's subsample to get the gist of the distribution.
x_generated = tf.reshape(tf.transpose(x_generated, [1, 0, 2]), (2, -1))[:, ::47]
plt.scatter(x_train[0, :], x_train[1, :], color='blue', alpha=0.1, label='Actual data')
plt.scatter(x_generated[0, :], x_generated[1, :], color='red', alpha=0.1, label='Simulated data (VI)')
plt.legend()
plt.axis([-20, 20, -20, 20])
plt.show()
Agradecimientos
Este tutorial fue escrito originalmente en Edward 1,0 ( fuente ). Agradecemos a todos los colaboradores por escribir y revisar esa versión.
Referencias
[1]: Michael E. Tipping y Christopher M. Bishop. Análisis probabilístico de componentes principales. Revista de la Sociedad Royal Statistical: Serie B (Metodología Estadística), 61 (3): 611-622, 1999.
