 TensorFlow.org এ দেখুন TensorFlow.org এ দেখুন |  Google Colab-এ চালান Google Colab-এ চালান |  GitHub-এ উৎস দেখুন GitHub-এ উৎস দেখুন |  নোটবুক ডাউনলোড করুন নোটবুক ডাউনলোড করুন |
import numpy as np
import matplotlib.pyplot as plt
import tensorflow.compat.v2 as tf
tf.enable_v2_behavior()
import tensorflow_probability as tfp
tfd = tfp.distributions
tfb = tfp.bijectors
একটি [যোজক পদ] (https://en.wikipedia.org/wiki/Copula_ (probability_theory% 29) র্যান্ডম ভেরিয়েবল মধ্যে নির্ভরতা ধরে রাখার জন্য একটি শাস্ত্রীয় পন্থা। আরো আনুষ্ঠানিকভাবে একটি যোজক একটি বহুচলকীয় বন্টন হয় \(C(U_1, U_2, ...., U_n)\) যেমন যে খর্ব করা দেয় \(U_i \sim \text{Uniform}(0, 1)\)।
কপুলাগুলি আকর্ষণীয় কারণ আমরা তাদের ব্যবহার করতে পারি নির্বিচারে প্রান্তিকের সাথে মাল্টিভেরিয়েট ডিস্ট্রিবিউশন তৈরি করতে। এই রেসিপি:
- ব্যবহার সম্ভাব্যতা সমাকলন ট্রান্সফর্ম পালাক্রমে একটি অবাধ একটানা আরভি \(X\) একটি অভিন্ন এক মধ্যে \(F_X(X)\), যেখানে \(F_X\) এর সিডিএফ হয় \(X\)।
- একটি যোজক (বলুন bivariate) দেওয়া \(C(U, V)\), আমরা যে আছে \(U\) এবং \(V\) অভিন্ন প্রান্তিক ডিস্ট্রিবিউশন আছে।
- এখন আমাদের আরভি সুদের এর দেওয়া \(X, Y\), একটি নতুন ডিস্ট্রিবিউশন নির্মাণের \(C'(X, Y) = C(F_X(X), F_Y(Y))\)। জন্য marginals \(X\) এবং \(Y\) বেশী আমরা আকাঙ্ক্ষিত হয়।
প্রান্তিকগুলি একক এবং এইভাবে পরিমাপ করা এবং/অথবা মডেল করা সহজ হতে পারে। একটি কপুলা প্রান্তিক থেকে শুরু করতে সক্ষম করে তবুও মাত্রার মধ্যে নির্বিচারে পারস্পরিক সম্পর্ক অর্জন করে।
গাউসিয়ান কপুলা
কপুলাগুলি কীভাবে তৈরি করা হয় তা বোঝাতে, মাল্টিভেরিয়েট গাউসিয়ান পারস্পরিক সম্পর্ক অনুসারে নির্ভরতা ক্যাপচার করার ক্ষেত্রে বিবেচনা করুন। একজন গসিয়ান যোজক পদ একের পর দেওয়া হয় \(C(u_1, u_2, ...u_n) = \Phi_\Sigma(\Phi^{-1}(u_1), \Phi^{-1}(u_2), ... \Phi^{-1}(u_n))\) যেখানে \(\Phi_\Sigma\) , একটি MultivariateNormal এর সিডিএফ প্রতিনিধিত্ব করে সহভেদাংক সঙ্গে \(\Sigma\) এবং গড় 0, এবং \(\Phi^{-1}\) আদর্শ স্বাভাবিক জন্য বিপরীত সিডিএফ হয়।
স্বাভাবিকের বিপরীত CDF প্রয়োগ করা সাধারণভাবে বিতরণ করা অভিন্ন মাত্রাগুলিকে বিকৃত করে। মাল্টিভেরিয়েট নরমালের CDF প্রয়োগ করলে বণ্টনকে সামান্য অভিন্ন এবং গাউসিয়ান পারস্পরিক সম্পর্ক সহ স্কোয়াশ করে।
সুতরাং, কি আমরা পেতে যে গসিয়ান যোজক পদ ইউনিট hypercube উপর একটি বিতরণ হয় \([0, 1]^n\) অভিন্ন marginals সঙ্গে।
যেমন সংজ্ঞায়িত গসিয়ান যোজক পদটির সঙ্গে বাস্তবায়ন করা যায় tfd.TransformedDistribution এবং উপযুক্ত Bijector । অর্থাৎ আমরা একটি MultivariateNormal রূপান্তর করা হয় সাধারন ডিস্ট্রিবিউশনের সিডিএফ বিপরীত ব্যবহার, দ্বারা বাস্তবায়িত মাধ্যমে tfb.NormalCDF bijector।
নীচে, আমরা এক সরল ধৃষ্টতা সঙ্গে একটি গসিয়ান যোজক পদ বাস্তবায়ন: যে সহভেদাংক একটি Cholesky ফ্যাক্টর (অত: পর একটি সহভেদাংক দ্বারা স্থিতিমাপ হয় MultivariateNormalTriL )। (অন্য একটি ব্যবহার করতে পারে tf.linalg.LinearOperators বিভিন্ন ম্যাট্রিক্স-মুক্ত অনুমানের সঙ্কেতাক্ষরে লিখা।)।
class GaussianCopulaTriL(tfd.TransformedDistribution):
"""Takes a location, and lower triangular matrix for the Cholesky factor."""
def __init__(self, loc, scale_tril):
super(GaussianCopulaTriL, self).__init__(
distribution=tfd.MultivariateNormalTriL(
loc=loc,
scale_tril=scale_tril),
bijector=tfb.NormalCDF(),
validate_args=False,
name="GaussianCopulaTriLUniform")
# Plot an example of this.
unit_interval = np.linspace(0.01, 0.99, num=200, dtype=np.float32)
x_grid, y_grid = np.meshgrid(unit_interval, unit_interval)
coordinates = np.concatenate(
[x_grid[..., np.newaxis],
y_grid[..., np.newaxis]], axis=-1)
pdf = GaussianCopulaTriL(
loc=[0., 0.],
scale_tril=[[1., 0.8], [0., 0.6]],
).prob(coordinates)
# Plot its density.
plt.contour(x_grid, y_grid, pdf, 100, cmap=plt.cm.jet);
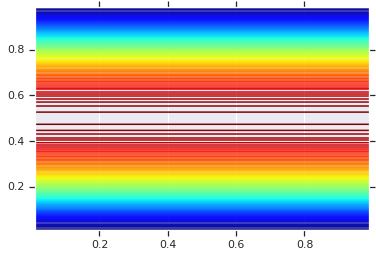
যাইহোক, এই ধরনের একটি মডেল থেকে পাওয়ারটি সম্ভাব্যতা ইন্টিগ্রাল ট্রান্সফর্ম ব্যবহার করে, নির্বিচারে আরভিতে কপুলা ব্যবহার করার জন্য এইভাবে, আমরা নির্বিচারে প্রান্তিকগুলি নির্দিষ্ট করতে পারি এবং তাদের একসাথে সেলাই করতে কপুলা ব্যবহার করতে পারি।
আমরা একটি মডেল দিয়ে শুরু করি:
\[\begin{align*} X &\sim \text{Kumaraswamy}(a, b) \\ Y &\sim \text{Gumbel}(\mu, \beta) \end{align*}\]
এবং যোজক পদ ব্যবহার bivariate আরভি পেতে \(Z\), যা marginals হয়েছে Kumaraswamy এবং Gumbel ।
আমরা সেই দুটি আরভি দ্বারা উত্পন্ন পণ্য বিতরণের প্লট করে শুরু করব এটি কেবলমাত্র আমরা যখন কপুলা প্রয়োগ করি তখন তুলনামূলক পয়েন্ট হিসাবে পরিবেশন করার জন্য।
a = 2.0
b = 2.0
gloc = 0.
gscale = 1.
x = tfd.Kumaraswamy(a, b)
y = tfd.Gumbel(loc=gloc, scale=gscale)
# Plot the distributions, assuming independence
x_axis_interval = np.linspace(0.01, 0.99, num=200, dtype=np.float32)
y_axis_interval = np.linspace(-2., 3., num=200, dtype=np.float32)
x_grid, y_grid = np.meshgrid(x_axis_interval, y_axis_interval)
pdf = x.prob(x_grid) * y.prob(y_grid)
# Plot its density
plt.contour(x_grid, y_grid, pdf, 100, cmap=plt.cm.jet);
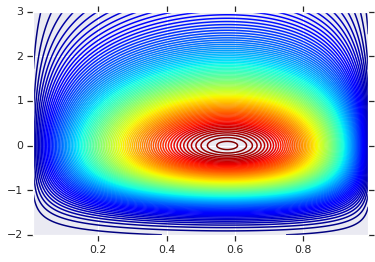
বিভিন্ন প্রান্তিকের সাথে যৌথ বন্টন
এখন আমরা একটি গাউসিয়ান কপুলা ব্যবহার করে বন্টনগুলিকে একত্রিত করতে এবং এটি প্লট করি। আবার পছন্দের আমাদের টুল TransformedDistribution উপযুক্ত প্রয়োগের Bijector মনোনীত marginals প্রাপ্ত।
বিশেষ করে, আমরা একটি ব্যবহার Blockwise (যা এখনও একটি bijective রূপান্তর হয়) ভেক্টর বিভিন্ন স্থানে বিভিন্ন bijectors প্রযোজ্য যা bijector।
এখন আমরা যে কপুলা চাই তা সংজ্ঞায়িত করতে পারি। লক্ষ্য প্রান্তিকের একটি তালিকা দেওয়া (বাইজেক্টর হিসাবে এনকোড করা), আমরা সহজেই একটি নতুন ডিস্ট্রিবিউশন তৈরি করতে পারি যা কপুলা ব্যবহার করে এবং নির্দিষ্ট প্রান্তিকগুলি রয়েছে।
class WarpedGaussianCopula(tfd.TransformedDistribution):
"""Application of a Gaussian Copula on a list of target marginals.
This implements an application of a Gaussian Copula. Given [x_0, ... x_n]
which are distributed marginally (with CDF) [F_0, ... F_n],
`GaussianCopula` represents an application of the Copula, such that the
resulting multivariate distribution has the above specified marginals.
The marginals are specified by `marginal_bijectors`: These are
bijectors whose `inverse` encodes the CDF and `forward` the inverse CDF.
block_sizes is a 1-D Tensor to determine splits for `marginal_bijectors`
length should be same as length of `marginal_bijectors`.
See tfb.Blockwise for details
"""
def __init__(self, loc, scale_tril, marginal_bijectors, block_sizes=None):
super(WarpedGaussianCopula, self).__init__(
distribution=GaussianCopulaTriL(loc=loc, scale_tril=scale_tril),
bijector=tfb.Blockwise(bijectors=marginal_bijectors,
block_sizes=block_sizes),
validate_args=False,
name="GaussianCopula")
অবশেষে, আসুন আসলে এই গাউসিয়ান কপুলা ব্যবহার করি। আমরা একটি Cholesky ব্যবহার করব \(\begin{bmatrix}1 & 0\\\rho & \sqrt{(1-\rho^2)}\end{bmatrix}\), যা 1 ভেরিয়ানস মিলা হবে, এবং পারস্পরিক সম্পর্ক \(\rho\) বহুচলকীয় স্বাভাবিক জন্য।
আমরা কয়েকটি ক্ষেত্রে দেখব:
# Create our coordinates:
coordinates = np.concatenate(
[x_grid[..., np.newaxis], y_grid[..., np.newaxis]], -1)
def create_gaussian_copula(correlation):
# Use Gaussian Copula to add dependence.
return WarpedGaussianCopula(
loc=[0., 0.],
scale_tril=[[1., 0.], [correlation, tf.sqrt(1. - correlation ** 2)]],
# These encode the marginals we want. In this case we want X_0 has
# Kumaraswamy marginal, and X_1 has Gumbel marginal.
marginal_bijectors=[
tfb.Invert(tfb.KumaraswamyCDF(a, b)),
tfb.Invert(tfb.GumbelCDF(loc=0., scale=1.))])
# Note that the zero case will correspond to independent marginals!
correlations = [0., -0.8, 0.8]
copulas = []
probs = []
for correlation in correlations:
copula = create_gaussian_copula(correlation)
copulas.append(copula)
probs.append(copula.prob(coordinates))
# Plot it's density
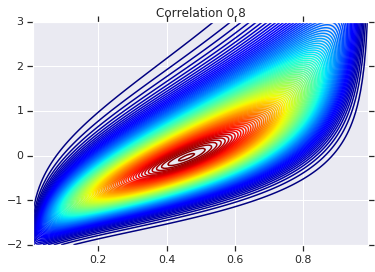
for correlation, copula_prob in zip(correlations, probs):
plt.figure()
plt.contour(x_grid, y_grid, copula_prob, 100, cmap=plt.cm.jet)
plt.title('Correlation {}'.format(correlation))
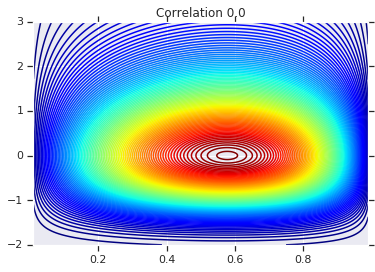
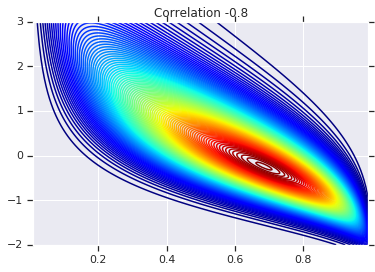
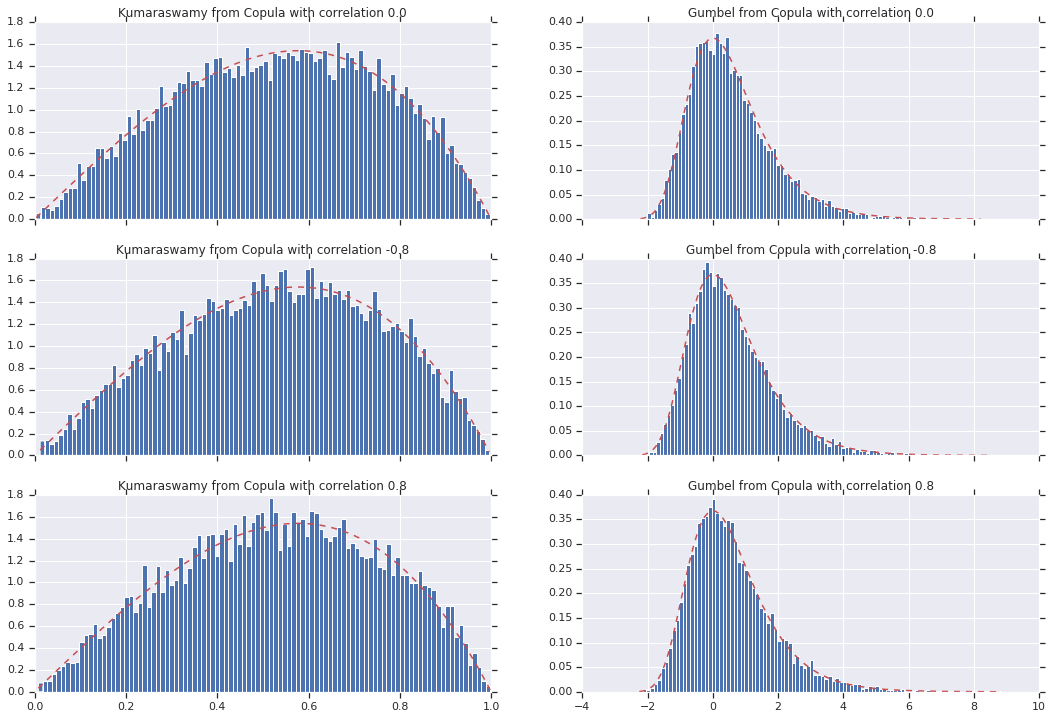
পরিশেষে, আসুন যাচাই করি যে আমরা আসলে আমরা যে প্রান্তিকগুলি চাই তা পাই।
def kumaraswamy_pdf(x):
return tfd.Kumaraswamy(a, b).prob(np.float32(x))
def gumbel_pdf(x):
return tfd.Gumbel(gloc, gscale).prob(np.float32(x))
copula_samples = []
for copula in copulas:
copula_samples.append(copula.sample(10000))
plot_rows = len(correlations)
plot_cols = 2 # for 2 densities [kumarswamy, gumbel]
fig, axes = plt.subplots(plot_rows, plot_cols, sharex='col', figsize=(18,12))
# Let's marginalize out on each, and plot the samples.
for i, (correlation, copula_sample) in enumerate(zip(correlations, copula_samples)):
k = copula_sample[..., 0].numpy()
g = copula_sample[..., 1].numpy()
_, bins, _ = axes[i, 0].hist(k, bins=100, density=True)
axes[i, 0].plot(bins, kumaraswamy_pdf(bins), 'r--')
axes[i, 0].set_title('Kumaraswamy from Copula with correlation {}'.format(correlation))
_, bins, _ = axes[i, 1].hist(g, bins=100, density=True)
axes[i, 1].plot(bins, gumbel_pdf(bins), 'r--')
axes[i, 1].set_title('Gumbel from Copula with correlation {}'.format(correlation))
উপসংহার
এবং আমরা সেখানে যেতে! আমরা প্রদর্শিত করেছি যে আমরা ব্যবহার গসিয়ান Copulas গঠন করা যেতে পারে Bijector API- টি।
আরো সাধারণভাবে, ব্যবহার bijectors লেখা Bijector এপিআই এবং তাদেরকে একটি ডিস্ট্রিবিউশনের সাথে রচনা নমনীয় মডেলিং জন্য ডিস্ট্রিবিউশন সমৃদ্ধ পরিবার তৈরি করতে পারেন।
