 Lihat di TensorFlow.org Lihat di TensorFlow.org |  Jalankan di Google Colab Jalankan di Google Colab |  Lihat sumber di GitHub Lihat sumber di GitHub |  Unduh buku catatan Unduh buku catatan |
Dalam colab ini kita akan mengeksplorasi pengambilan sampel dari bagian belakang Model Campuran Gaussian Bayesian (BGMM) hanya menggunakan primitif TensorFlow Probability.
Model
Untuk \(k\in\{1,\ldots, K\}\) komponen campuran masing-masing dimensi \(D\), kami ingin Model \(i\in\{1,\ldots,N\}\) sampel menggunakan berikut Bayesian Gaussian Mixture Model iid:
\[\begin{align*} \theta &\sim \text{Dirichlet}(\text{concentration}=\alpha_0)\\ \mu_k &\sim \text{Normal}(\text{loc}=\mu_{0k}, \text{scale}=I_D)\\ T_k &\sim \text{Wishart}(\text{df}=5, \text{scale}=I_D)\\ Z_i &\sim \text{Categorical}(\text{probs}=\theta)\\ Y_i &\sim \text{Normal}(\text{loc}=\mu_{z_i}, \text{scale}=T_{z_i}^{-1/2})\\ \end{align*}\]
Catatan, scale argumen semua memiliki cholesky semantik. Kami menggunakan konvensi ini karena ini adalah TF Distributions (yang sendiri menggunakan konvensi ini sebagian karena menguntungkan secara komputasi).
Tujuan kami adalah untuk menghasilkan sampel dari posterior:
\[p\left(\theta, \{\mu_k, T_k\}_{k=1}^K \Big| \{y_i\}_{i=1}^N, \alpha_0, \{\mu_{ok}\}_{k=1}^K\right)\]
Perhatikan bahwa \(\{Z_i\}_{i=1}^N\) tidak hadir - kita tertarik hanya mereka variabel acak yang tidak skala dengan \(N\). (Dan untungnya ada distribusi TF yang menangani meminggirkan keluar \(Z_i\).)
Tidak mungkin untuk mengambil sampel secara langsung dari distribusi ini karena istilah normalisasi yang sulit secara komputasional.
Algoritma Metropolis-Hastings yang teknik untuk untuk pengambilan sampel dari distribusi keras-to-menormalkan.
TensorFlow Probability menawarkan sejumlah opsi MCMC, termasuk beberapa berdasarkan Metropolis-Hastings. Dalam notebook ini, kita akan menggunakan Hamiltonian Monte Carlo ( tfp.mcmc.HamiltonianMonteCarlo ). HMC seringkali merupakan pilihan yang baik karena dapat menyatu dengan cepat, mengambil sampel ruang keadaan bersama-sama (sebagai lawan dari koordinat), dan memanfaatkan salah satu keunggulan TF: diferensiasi otomatis. Yang mengatakan, sampel dari posterior BGMM mungkin sebenarnya lebih baik dilakukan oleh pendekatan-pendekatan lain, misalnya, pengambilan sampel Gibb .
%matplotlib inline
import functools
import matplotlib.pyplot as plt; plt.style.use('ggplot')
import numpy as np
import seaborn as sns; sns.set_context('notebook')
import tensorflow.compat.v2 as tf
tf.enable_v2_behavior()
import tensorflow_probability as tfp
tfd = tfp.distributions
tfb = tfp.bijectors
physical_devices = tf.config.experimental.list_physical_devices('GPU')
if len(physical_devices) > 0:
tf.config.experimental.set_memory_growth(physical_devices[0], True)
Sebelum benar-benar membangun model, kita perlu mendefinisikan tipe distribusi baru. Dari spesifikasi model di atas, jelas kita sedang membuat parameter MVN dengan matriks kovarians terbalik, yaitu [matriks presisi](https://en.wikipedia.org/wiki/Precision_(statistics%29). TF, kita harus menggelar kami Bijector ini. Bijector akan menggunakan transformasi ke depan:
-
Y = tf.linalg.triangular_solve((tf.linalg.matrix_transpose(chol_precision_tril), X, adjoint=True) + loc.
Dan log_prob perhitungan hanya kebalikan, yaitu:
-
X = tf.linalg.matmul(chol_precision_tril, X - loc, adjoint_a=True).
Karena semua kita perlu untuk HMC adalah log_prob , ini berarti kita menghindari pernah memanggil tf.linalg.triangular_solve (seperti yang akan menjadi kasus untuk tfd.MultivariateNormalTriL ). Hal ini menguntungkan karena tf.linalg.matmul biasanya lebih cepat karena lokalitas cache yang lebih baik.
class MVNCholPrecisionTriL(tfd.TransformedDistribution):
"""MVN from loc and (Cholesky) precision matrix."""
def __init__(self, loc, chol_precision_tril, name=None):
super(MVNCholPrecisionTriL, self).__init__(
distribution=tfd.Independent(tfd.Normal(tf.zeros_like(loc),
scale=tf.ones_like(loc)),
reinterpreted_batch_ndims=1),
bijector=tfb.Chain([
tfb.Shift(shift=loc),
tfb.Invert(tfb.ScaleMatvecTriL(scale_tril=chol_precision_tril,
adjoint=True)),
]),
name=name)
The tfd.Independent bergantian distribusi independen menarik dari satu distribusi, menjadi distribusi multivariat dengan koordinat statistik independen. Dalam hal komputasi log_prob , ini "meta-distribusi" bermanifestasi sebagai penjumlahan sederhana atas dimensi acara (s).
Juga perhatikan bahwa kami mengambil adjoint ( "transpose") dari matriks skala. Hal ini karena jika presisi adalah kovarians terbalik, yaitu, \(P=C^{-1}\) dan jika \(C=AA^\top\), maka \(P=BB^{\top}\) mana \(B=A^{-\top}\).
Karena distribusi ini agak sulit, mari kita cepat memverifikasi bahwa kami MVNCholPrecisionTriL bekerja seperti yang kita pikir seharusnya.
def compute_sample_stats(d, seed=42, n=int(1e6)):
x = d.sample(n, seed=seed)
sample_mean = tf.reduce_mean(x, axis=0, keepdims=True)
s = x - sample_mean
sample_cov = tf.linalg.matmul(s, s, adjoint_a=True) / tf.cast(n, s.dtype)
sample_scale = tf.linalg.cholesky(sample_cov)
sample_mean = sample_mean[0]
return [
sample_mean,
sample_cov,
sample_scale,
]
dtype = np.float32
true_loc = np.array([1., -1.], dtype=dtype)
true_chol_precision = np.array([[1., 0.],
[2., 8.]],
dtype=dtype)
true_precision = np.matmul(true_chol_precision, true_chol_precision.T)
true_cov = np.linalg.inv(true_precision)
d = MVNCholPrecisionTriL(
loc=true_loc,
chol_precision_tril=true_chol_precision)
[sample_mean, sample_cov, sample_scale] = [
t.numpy() for t in compute_sample_stats(d)]
print('true mean:', true_loc)
print('sample mean:', sample_mean)
print('true cov:\n', true_cov)
print('sample cov:\n', sample_cov)
true mean: [ 1. -1.] sample mean: [ 1.0002806 -1.000105 ] true cov: [[ 1.0625 -0.03125 ] [-0.03125 0.015625]] sample cov: [[ 1.0641273 -0.03126175] [-0.03126175 0.01559312]]
Karena rata-rata sampel dan kovarians mendekati rata-rata dan kovarians sebenarnya, sepertinya distribusi diimplementasikan dengan benar. Sekarang, kita akan menggunakan MVNCholPrecisionTriL tfp.distributions.JointDistributionNamed untuk menentukan model BGMM. Untuk model observasional, kita akan menggunakan tfd.MixtureSameFamily untuk secara otomatis mengintegrasikan keluar \(\{Z_i\}_{i=1}^N\) menarik.
dtype = np.float64
dims = 2
components = 3
num_samples = 1000
bgmm = tfd.JointDistributionNamed(dict(
mix_probs=tfd.Dirichlet(
concentration=np.ones(components, dtype) / 10.),
loc=tfd.Independent(
tfd.Normal(
loc=np.stack([
-np.ones(dims, dtype),
np.zeros(dims, dtype),
np.ones(dims, dtype),
]),
scale=tf.ones([components, dims], dtype)),
reinterpreted_batch_ndims=2),
precision=tfd.Independent(
tfd.WishartTriL(
df=5,
scale_tril=np.stack([np.eye(dims, dtype=dtype)]*components),
input_output_cholesky=True),
reinterpreted_batch_ndims=1),
s=lambda mix_probs, loc, precision: tfd.Sample(tfd.MixtureSameFamily(
mixture_distribution=tfd.Categorical(probs=mix_probs),
components_distribution=MVNCholPrecisionTriL(
loc=loc,
chol_precision_tril=precision)),
sample_shape=num_samples)
))
def joint_log_prob(observations, mix_probs, loc, chol_precision):
"""BGMM with priors: loc=Normal, precision=Inverse-Wishart, mix=Dirichlet.
Args:
observations: `[n, d]`-shaped `Tensor` representing Bayesian Gaussian
Mixture model draws. Each sample is a length-`d` vector.
mix_probs: `[K]`-shaped `Tensor` representing random draw from
`Dirichlet` prior.
loc: `[K, d]`-shaped `Tensor` representing the location parameter of the
`K` components.
chol_precision: `[K, d, d]`-shaped `Tensor` representing `K` lower
triangular `cholesky(Precision)` matrices, each being sampled from
a Wishart distribution.
Returns:
log_prob: `Tensor` representing joint log-density over all inputs.
"""
return bgmm.log_prob(
mix_probs=mix_probs, loc=loc, precision=chol_precision, s=observations)
Hasilkan Data "Pelatihan"
Untuk demo ini, kami akan mengambil sampel beberapa data acak.
true_loc = np.array([[-2., -2],
[0, 0],
[2, 2]], dtype)
random = np.random.RandomState(seed=43)
true_hidden_component = random.randint(0, components, num_samples)
observations = (true_loc[true_hidden_component] +
random.randn(num_samples, dims).astype(dtype))
Inferensi Bayesian menggunakan HMC
Sekarang kami telah menggunakan TFD untuk menentukan model kami dan memperoleh beberapa data yang diamati, kami memiliki semua bagian yang diperlukan untuk menjalankan HMC.
Untuk melakukan hal ini, kita akan menggunakan aplikasi parsial untuk "pin down" hal yang kita tidak ingin sampel. Dalam hal ini yang berarti kita hanya perlu dijabarkan observations . (The hiper-parameter sudah dipanggang untuk distribusi sebelumnya dan bukan bagian dari joint_log_prob fungsi tanda tangan.)
unnormalized_posterior_log_prob = functools.partial(joint_log_prob, observations)
initial_state = [
tf.fill([components],
value=np.array(1. / components, dtype),
name='mix_probs'),
tf.constant(np.array([[-2., -2],
[0, 0],
[2, 2]], dtype),
name='loc'),
tf.linalg.eye(dims, batch_shape=[components], dtype=dtype, name='chol_precision'),
]
Representasi Tanpa Batas
Hamiltonian Monte Carlo (HMC) membutuhkan fungsi log-probabilitas target yang dapat didiferensiasikan sehubungan dengan argumennya. Selanjutnya, HMC dapat menunjukkan efisiensi statistik yang lebih tinggi secara dramatis jika ruang keadaan tidak dibatasi.
Ini berarti kita harus menyelesaikan dua masalah utama saat mengambil sampel dari posterior BGMM:
- \(\theta\) merupakan vektor probabilitas diskrit, yaitu, harus sedemikian rupa sehingga \(\sum_{k=1}^K \theta_k = 1\) dan \(\theta_k>0\).
- \(T_k\) merupakan inverse kovarians matriks, yaitu, harus sedemikian rupa sehingga \(T_k \succ 0\), yaitu, adalah definit positif .
Untuk mengatasi persyaratan ini, kita perlu:
- mengubah variabel dibatasi ke ruang tak terbatas
- jalankan MCMC di ruang yang tidak dibatasi
- mengubah variabel yang tidak dibatasi kembali ke ruang terbatas.
Seperti MVNCholPrecisionTriL , kita akan menggunakan Bijector s untuk mengubah variabel acak ruang dibatasi.
The
Dirichletditransformasikan ke ruang dibatasi melalui fungsi Softmax .Variabel acak presisi kami adalah distribusi di atas matriks semidefinite postive. Untuk hapuslah kendala ini kita akan menggunakan
FillTriangulardanTransformDiagonalbijectors. Ini mengubah vektor menjadi matriks segitiga bawah dan memastikan diagonalnya positif. Mantan berguna karena memungkinkan pengambilan sampel hanya \(d(d+1)/2\) mengapung daripada \(d^2\).
unconstraining_bijectors = [
tfb.SoftmaxCentered(),
tfb.Identity(),
tfb.Chain([
tfb.TransformDiagonal(tfb.Softplus()),
tfb.FillTriangular(),
])]
@tf.function(autograph=False)
def sample():
return tfp.mcmc.sample_chain(
num_results=2000,
num_burnin_steps=500,
current_state=initial_state,
kernel=tfp.mcmc.SimpleStepSizeAdaptation(
tfp.mcmc.TransformedTransitionKernel(
inner_kernel=tfp.mcmc.HamiltonianMonteCarlo(
target_log_prob_fn=unnormalized_posterior_log_prob,
step_size=0.065,
num_leapfrog_steps=5),
bijector=unconstraining_bijectors),
num_adaptation_steps=400),
trace_fn=lambda _, pkr: pkr.inner_results.inner_results.is_accepted)
[mix_probs, loc, chol_precision], is_accepted = sample()
Kami sekarang akan mengeksekusi rantai dan mencetak sarana posterior.
acceptance_rate = tf.reduce_mean(tf.cast(is_accepted, dtype=tf.float32)).numpy()
mean_mix_probs = tf.reduce_mean(mix_probs, axis=0).numpy()
mean_loc = tf.reduce_mean(loc, axis=0).numpy()
mean_chol_precision = tf.reduce_mean(chol_precision, axis=0).numpy()
precision = tf.linalg.matmul(chol_precision, chol_precision, transpose_b=True)
print('acceptance_rate:', acceptance_rate)
print('avg mix probs:', mean_mix_probs)
print('avg loc:\n', mean_loc)
print('avg chol(precision):\n', mean_chol_precision)
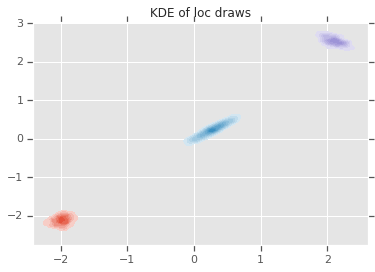
acceptance_rate: 0.5305 avg mix probs: [0.25248723 0.60729516 0.1402176 ] avg loc: [[-1.96466753 -2.12047249] [ 0.27628865 0.22944732] [ 2.06461244 2.54216122]] avg chol(precision): [[[ 1.05105032 0. ] [ 0.12699955 1.06553113]] [[ 0.76058015 0. ] [-0.50332767 0.77947431]] [[ 1.22770457 0. ] [ 0.70670027 1.50914164]]]
loc_ = loc.numpy()
ax = sns.kdeplot(loc_[:,0,0], loc_[:,0,1], shade=True, shade_lowest=False)
ax = sns.kdeplot(loc_[:,1,0], loc_[:,1,1], shade=True, shade_lowest=False)
ax = sns.kdeplot(loc_[:,2,0], loc_[:,2,1], shade=True, shade_lowest=False)
plt.title('KDE of loc draws');
Kesimpulan
Colab sederhana ini menunjukkan bagaimana primitif TensorFlow Probability dapat digunakan untuk membangun model campuran Bayesian hierarkis.
