- Descrizione :
'ogbg-molpcba' è un set di dati molecolari campionato da PubChem BioAssay. È un set di dati di previsione del grafico dall'Open Graph Benchmark (OGB).
Questo set di dati è sperimentale e l'API è soggetta a modifiche nelle versioni future.
La seguente descrizione del set di dati è adattata dal documento OGB:
Formato di input
Tutte le molecole sono pre-elaborate utilizzando RDKit ([1]).
- Ogni grafico rappresenta una molecola, dove i nodi sono atomi e i bordi sono legami chimici.
- Le caratteristiche del nodo di input sono a 9 dimensioni, contenenti il numero atomico e la chiralità, così come altre caratteristiche atomiche aggiuntive come la carica formale e se l'atomo è nell'anello.
- Le caratteristiche del bordo di input sono tridimensionali e contengono il tipo di legame, la stereochimica del legame, nonché una caratteristica aggiuntiva del legame che indica se il legame è coniugato.
La descrizione esatta di tutte le funzionalità è disponibile su https://github.com/snap-stanford/ogb/blob/master/ogb/utils/features.py
Predizione
Il compito è prevedere 128 diverse attività biologiche (inattive/attive). Vedere [2] e [3] per ulteriori descrizioni su questi obiettivi. Non tutti i bersagli si applicano a ciascuna molecola: i bersagli mancanti sono indicati dai NaN.
Riferimenti
[1]: Greg Landrum, et al. "RDKit: chemioinformatica open source". URL: https://github.com/rdkit/rdkit
[2]: Bharath Ramsundar, Steven Kearnes, Patrick Riley, Dale Webster, David Konerding e Vijay Pande. "Reti massicciamente multitasking per la scoperta di farmaci". URL: https://arxiv.org/pdf/1502.02072.pdf
[3]: Zhenqin Wu, Bharath Ramsundar, Evan N Feinberg, Joseph Gomes, Caleb Geniesse, Aneesh S. Pappu, Karl Leswing e Vijay Pande. MoleculeNet: un punto di riferimento per il machine learning molecolare. Scienze chimiche, 9(2):513-530, 2018.
Pagina iniziale : https://ogb.stanford.edu/docs/graphprop
Codice sorgente :
tfds.datasets.ogbg_molpcba.BuilderVersioni :
-
0.1.0: rilascio iniziale dell'API sperimentale. -
0.1.1: espone in modo esplicito il numero di spigoli in ciascun grafico. -
0.1.2: Aggiunta di un campo di metadati per GraphVisualizer. -
0.1.3(predefinito): aggiungere un campo di metadati per i nomi delle singole attività.
-
Dimensione del download :
37.70 MiBDimensione del set di dati:
822.53 MiBCache automatica ( documentazione ): No
Divisioni :
| Diviso | Esempi |
|---|---|
'test' | 43.793 |
'train' | 350.343 |
'validation' | 43.793 |
- Struttura delle caratteristiche :
FeaturesDict({
'edge_feat': Tensor(shape=(None, 3), dtype=float32),
'edge_index': Tensor(shape=(None, 2), dtype=int64),
'labels': Tensor(shape=(128,), dtype=float32),
'node_feat': Tensor(shape=(None, 9), dtype=float32),
'num_edges': Tensor(shape=(None,), dtype=int64),
'num_nodes': Tensor(shape=(None,), dtype=int64),
})
- Documentazione delle funzionalità :
| Caratteristica | Classe | Forma | Tipo D | Descrizione |
|---|---|---|---|---|
| CaratteristicheDict | ||||
| edge_feat | Tensore | (Nessuno, 3) | galleggiante32 | |
| bordo_indice | Tensore | (Nessuno, 2) | int64 | |
| etichette | Tensore | (128,) | galleggiante32 | |
| node_feat | Tensore | (Nessuno, 9) | galleggiante32 | |
| num_bordi | Tensore | (Nessuno,) | int64 | |
| num_nodi | Tensore | (Nessuno,) | int64 |
Chiavi supervisionate (Vedi
as_superviseddoc ):NoneFigura ( tfds.show_examples ):
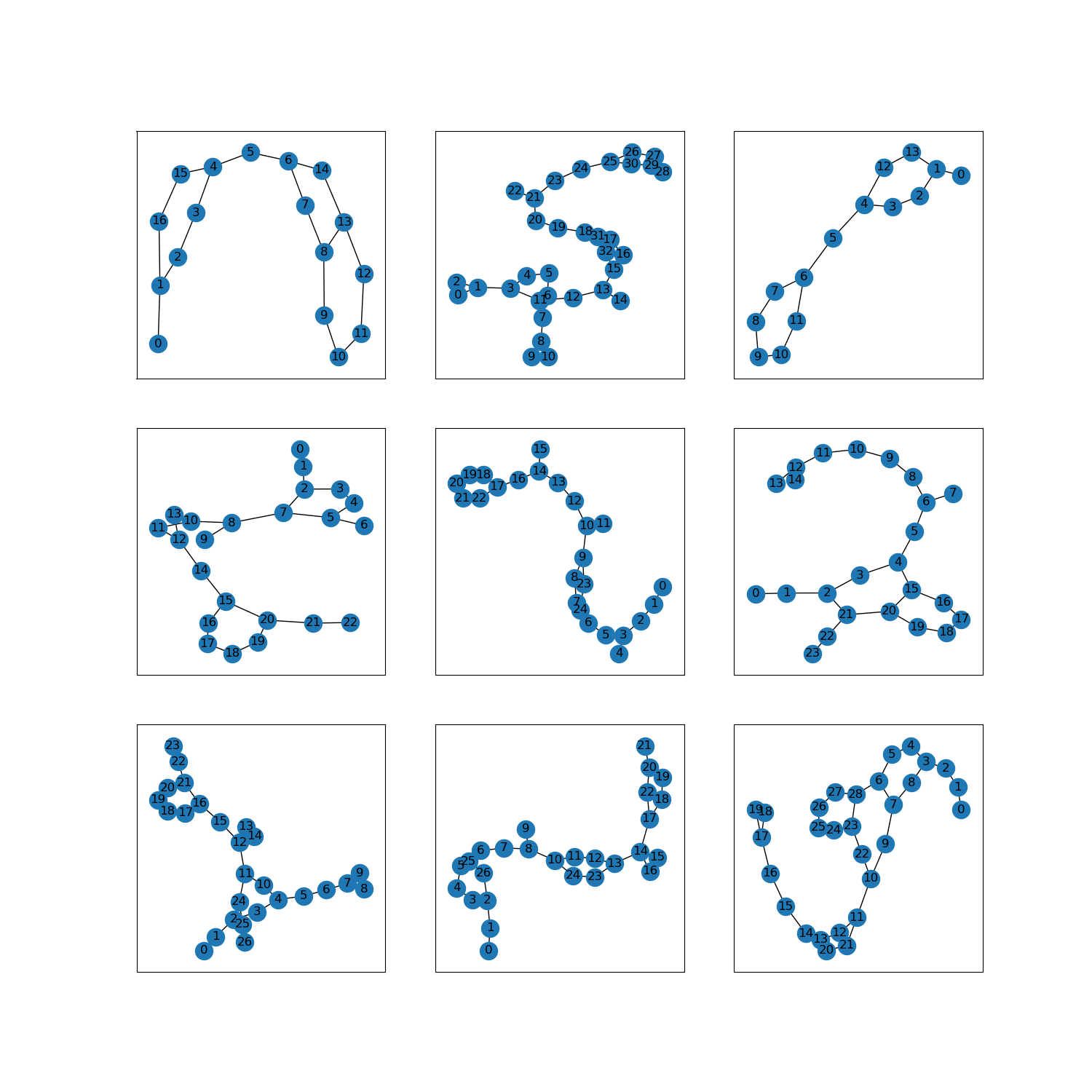
- Esempi ( tfds.as_dataframe ):
- Citazione :
@inproceedings{DBLP:conf/nips/HuFZDRLCL20,
author = {Weihua Hu and
Matthias Fey and
Marinka Zitnik and
Yuxiao Dong and
Hongyu Ren and
Bowen Liu and
Michele Catasta and
Jure Leskovec},
editor = {Hugo Larochelle and
Marc Aurelio Ranzato and
Raia Hadsell and
Maria{-}Florina Balcan and
Hsuan{-}Tien Lin},
title = {Open Graph Benchmark: Datasets for Machine Learning on Graphs},
booktitle = {Advances in Neural Information Processing Systems 33: Annual Conference
on Neural Information Processing Systems 2020, NeurIPS 2020, December
6-12, 2020, virtual},
year = {2020},
url = {https://proceedings.neurips.cc/paper/2020/hash/fb60d411a5c5b72b2e7d3527cfc84fd0-Abstract.html},
timestamp = {Tue, 19 Jan 2021 15:57:06 +0100},
biburl = {https://dblp.org/rec/conf/nips/HuFZDRLCL20.bib},
bibsource = {dblp computer science bibliography, https://dblp.org}
}

