- Descripción :
'ogbg-molpcba' es un conjunto de datos moleculares extraído de PubChem BioAssay. Es un conjunto de datos de predicción de gráficos de Open Graph Benchmark (OGB).
Este conjunto de datos es experimental y la API está sujeta a cambios en versiones futuras.
La siguiente descripción del conjunto de datos está adaptada del documento OGB:
Formato de entrada
Todas las moléculas se preprocesan utilizando RDKit ([1]).
- Cada gráfico representa una molécula, donde los nodos son átomos y los bordes son enlaces químicos.
- Las características del nodo de entrada son de 9 dimensiones y contienen el número atómico y la quiralidad, así como otras características atómicas adicionales, como la carga formal y si el átomo está en el anillo.
- Las características de borde de entrada son tridimensionales, contienen tipo de enlace, estereoquímica de enlace, así como una característica de enlace adicional que indica si el enlace está conjugado.
La descripción exacta de todas las funciones está disponible en https://github.com/snap-stanford/ogb/blob/master/ogb/utils/features.py
Predicción
La tarea es predecir 128 actividades biológicas diferentes (inactivas/activas). Consulte [2] y [3] para obtener más información sobre estos objetivos. No todos los objetivos se aplican a cada molécula: los NaN indican los objetivos que faltan.
Referencias
[1]: Greg Landrum, et al. 'RDKit: quimioinformática de código abierto'. URL: https://github.com/rdkit/rdkit
[2]: Bharath Ramsundar, Steven Kearnes, Patrick Riley, Dale Webster, David Konerding y Vijay Pande. 'Redes masivamente multitarea para el descubrimiento de fármacos'. URL: https://arxiv.org/pdf/1502.02072.pdf
[3]: Zhenqin Wu, Bharath Ramsundar, Evan N Feinberg, Joseph Gomes, Caleb Geniesse, Aneesh S. Pappu, Karl Leswing y Vijay Pande. MoleculeNet: un punto de referencia para el aprendizaje automático molecular. Ciencias químicas, 9(2):513-530, 2018.
Página de inicio: https://ogb.stanford.edu/docs/graphprop
Código fuente :
tfds.datasets.ogbg_molpcba.BuilderVersiones :
-
0.1.0: versión inicial de la API experimental. -
0.1.1: Expone el número de aristas en cada gráfico de forma explícita. -
0.1.2: Agregar campo de metadatos para GraphVisualizer. -
0.1.3(predeterminado): agregue un campo de metadatos para los nombres de tareas individuales.
-
Tamaño de la descarga :
37.70 MiBTamaño del conjunto de datos :
822.53 MiBAlmacenamiento automático en caché ( documentación ): No
Divisiones :
| Dividir | Ejemplos |
|---|---|
'test' | 43,793 |
'train' | 350,343 |
'validation' | 43,793 |
- Estructura de características :
FeaturesDict({
'edge_feat': Tensor(shape=(None, 3), dtype=float32),
'edge_index': Tensor(shape=(None, 2), dtype=int64),
'labels': Tensor(shape=(128,), dtype=float32),
'node_feat': Tensor(shape=(None, 9), dtype=float32),
'num_edges': Tensor(shape=(None,), dtype=int64),
'num_nodes': Tensor(shape=(None,), dtype=int64),
})
- Documentación de características :
| Característica | Clase | Forma | Tipo D | Descripción |
|---|---|---|---|---|
| CaracterísticasDict | ||||
| edge_feat | Tensor | (Ninguno, 3) | flotar32 | |
| índice_de_borde | Tensor | (Ninguno, 2) | int64 | |
| etiquetas | Tensor | (128,) | flotar32 | |
| node_feat | Tensor | (Ninguno, 9) | flotar32 | |
| num_bordes | Tensor | (Ninguno,) | int64 | |
| núm_nodos | Tensor | (Ninguno,) | int64 |
Claves supervisadas (Ver
as_superviseddoc ):NoneFigura ( tfds.show_examples ):
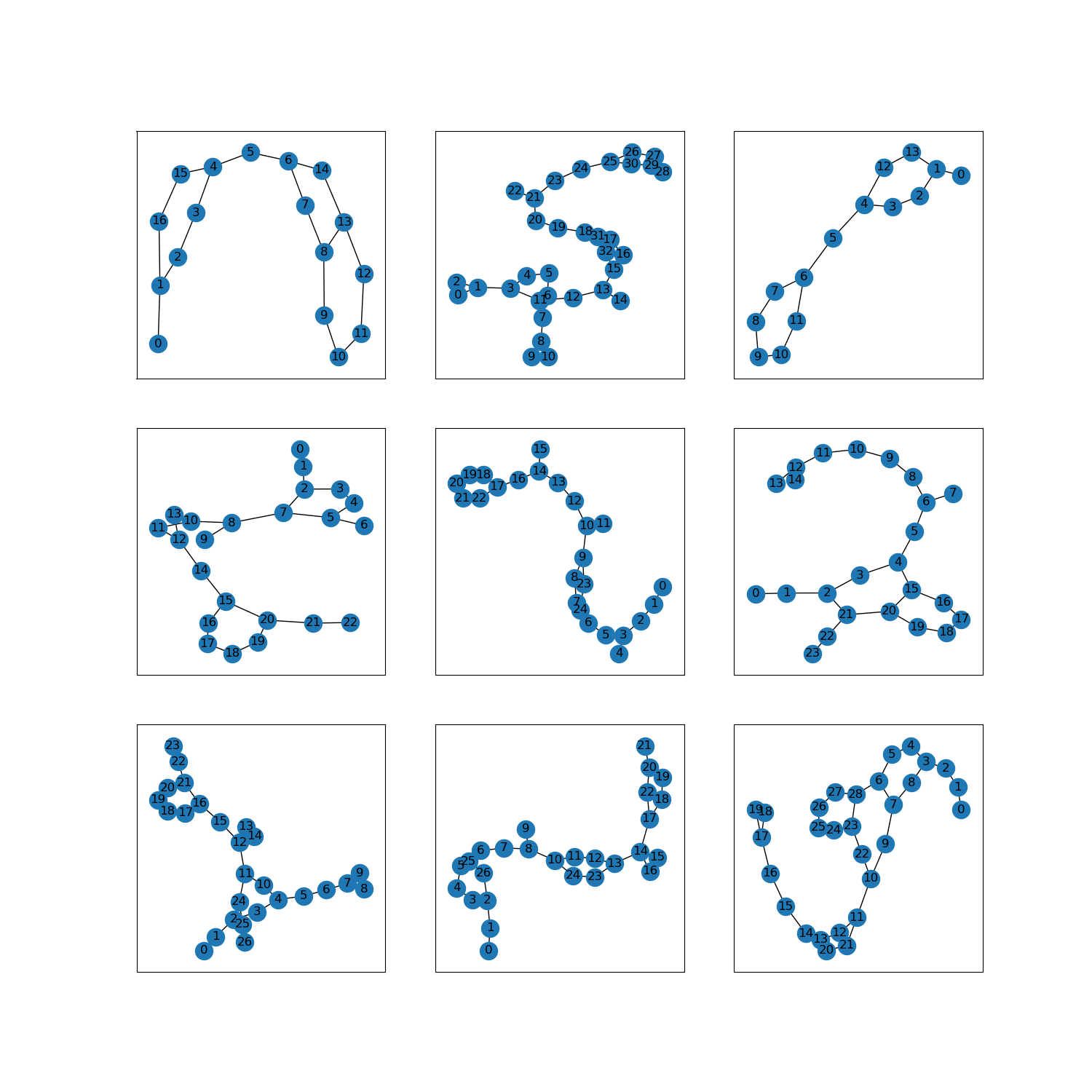
- Ejemplos ( tfds.as_dataframe ):
- Cita :
@inproceedings{DBLP:conf/nips/HuFZDRLCL20,
author = {Weihua Hu and
Matthias Fey and
Marinka Zitnik and
Yuxiao Dong and
Hongyu Ren and
Bowen Liu and
Michele Catasta and
Jure Leskovec},
editor = {Hugo Larochelle and
Marc Aurelio Ranzato and
Raia Hadsell and
Maria{-}Florina Balcan and
Hsuan{-}Tien Lin},
title = {Open Graph Benchmark: Datasets for Machine Learning on Graphs},
booktitle = {Advances in Neural Information Processing Systems 33: Annual Conference
on Neural Information Processing Systems 2020, NeurIPS 2020, December
6-12, 2020, virtual},
year = {2020},
url = {https://proceedings.neurips.cc/paper/2020/hash/fb60d411a5c5b72b2e7d3527cfc84fd0-Abstract.html},
timestamp = {Tue, 19 Jan 2021 15:57:06 +0100},
biburl = {https://dblp.org/rec/conf/nips/HuFZDRLCL20.bib},
bibsource = {dblp computer science bibliography, https://dblp.org}
}

