- Descriptif :
'ogbg-molpcba' est un ensemble de données moléculaires échantillonnées à partir de PubChem BioAssay. Il s'agit d'un ensemble de données de prédiction de graphe de l'Open Graph Benchmark (OGB).
Cet ensemble de données est expérimental et l'API est susceptible d'être modifiée dans les versions futures.
La description ci-dessous de l'ensemble de données est adaptée de l'article OGB :
Format d'entrée
Toutes les molécules sont pré-traitées à l'aide de RDKit ([1]).
- Chaque graphique représente une molécule, où les nœuds sont des atomes et les arêtes sont des liaisons chimiques.
- Les caractéristiques des nœuds d'entrée sont à 9 dimensions, contenant le numéro atomique et la chiralité, ainsi que d'autres caractéristiques atomiques supplémentaires telles que la charge formelle et si l'atome est dans l'anneau.
- Les caractéristiques de bord d'entrée sont tridimensionnelles, contenant le type de liaison, la stéréochimie de liaison, ainsi qu'une caractéristique de liaison supplémentaire indiquant si la liaison est conjuguée.
La description exacte de toutes les fonctionnalités est disponible sur https://github.com/snap-stanford/ogb/blob/master/ogb/utils/features.py
Prédiction
La tâche consiste à prédire 128 activités biologiques différentes (inactives/actives). Voir [2] et [3] pour plus de description sur ces cibles. Toutes les cibles ne s'appliquent pas à chaque molécule : les cibles manquantes sont indiquées par des NaN.
Les références
[1] : Greg Landrum, et al. 'RDKit: Cheminformatics open-source'. URL : https://github.com/rdkit/rdkit
[2] : Bharath Ramsundar, Steven Kearnes, Patrick Riley, Dale Webster, David Konerding et Vijay Pande. "Réseaux massivement multitâches pour la découverte de médicaments". URL : https://arxiv.org/pdf/1502.02072.pdf
[3] : Zhenqin Wu, Bharath Ramsundar, Evan N Feinberg, Joseph Gomes, Caleb Geniesse, Aneesh S. Pappu, Karl Leswing et Vijay Pande. MoleculeNet : une référence pour l'apprentissage automatique moléculaire. Science chimique, 9(2):513-530, 2018.
Page d' accueil : https://ogb.stanford.edu/docs/graphprop
Code source :
tfds.datasets.ogbg_molpcba.BuilderVersions :
-
0.1.0: Version initiale de l'API expérimentale. -
0.1.1: Expose explicitement le nombre d'arêtes dans chaque graphe. -
0.1.2: Ajout d'un champ de métadonnées pour GraphVisualizer. -
0.1.3(par défaut) : Ajouter un champ de métadonnées pour les noms des tâches individuelles.
-
Taille du téléchargement :
37.70 MiBTaille du jeu de données :
822.53 MiBMise en cache automatique ( documentation ): Non
Fractionnements :
| Diviser | Exemples |
|---|---|
'test' | 43 793 |
'train' | 350 343 |
'validation' | 43 793 |
- Structure des fonctionnalités :
FeaturesDict({
'edge_feat': Tensor(shape=(None, 3), dtype=float32),
'edge_index': Tensor(shape=(None, 2), dtype=int64),
'labels': Tensor(shape=(128,), dtype=float32),
'node_feat': Tensor(shape=(None, 9), dtype=float32),
'num_edges': Tensor(shape=(None,), dtype=int64),
'num_nodes': Tensor(shape=(None,), dtype=int64),
})
- Documentation des fonctionnalités :
| Fonctionnalité | Classe | Forme | Dtype | Description |
|---|---|---|---|---|
| FonctionnalitésDict | ||||
| bord_feat | Tenseur | (Aucun, 3) | float32 | |
| edge_index | Tenseur | (Aucun, 2) | int64 | |
| Étiquettes | Tenseur | (128,) | float32 | |
| node_feat | Tenseur | (Aucun, 9) | float32 | |
| num_edges | Tenseur | (Aucun,) | int64 | |
| num_nodes | Tenseur | (Aucun,) | int64 |
Clés supervisées (Voir
as_superviseddoc ):NoneFigure ( tfds.show_examples ):
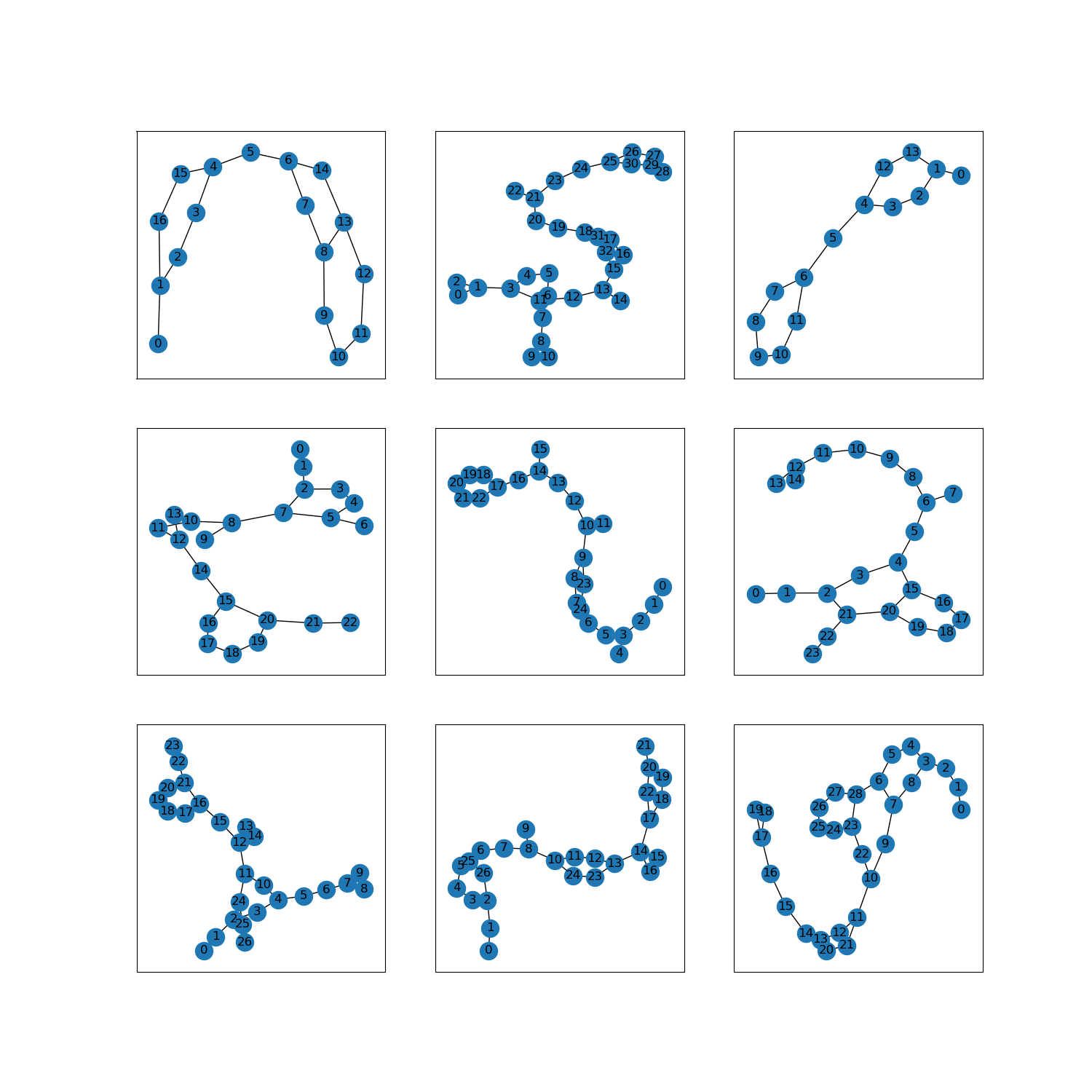
- Exemples ( tfds.as_dataframe ):
- Citation :
@inproceedings{DBLP:conf/nips/HuFZDRLCL20,
author = {Weihua Hu and
Matthias Fey and
Marinka Zitnik and
Yuxiao Dong and
Hongyu Ren and
Bowen Liu and
Michele Catasta and
Jure Leskovec},
editor = {Hugo Larochelle and
Marc Aurelio Ranzato and
Raia Hadsell and
Maria{-}Florina Balcan and
Hsuan{-}Tien Lin},
title = {Open Graph Benchmark: Datasets for Machine Learning on Graphs},
booktitle = {Advances in Neural Information Processing Systems 33: Annual Conference
on Neural Information Processing Systems 2020, NeurIPS 2020, December
6-12, 2020, virtual},
year = {2020},
url = {https://proceedings.neurips.cc/paper/2020/hash/fb60d411a5c5b72b2e7d3527cfc84fd0-Abstract.html},
timestamp = {Tue, 19 Jan 2021 15:57:06 +0100},
biburl = {https://dblp.org/rec/conf/nips/HuFZDRLCL20.bib},
bibsource = {dblp computer science bibliography, https://dblp.org}
}

