- Descrizione :
Il CBIS-DDSM (Curated Breast Imaging Subset of DDSM) è una versione aggiornata e standardizzata del Digital Database for Screening Mammography (DDSM). Il DDSM è un database di 2.620 studi mammografici su pellicola scansionata. Contiene casi normali, benigni e maligni con informazioni patologiche verificate.
La configurazione predefinita è composta da patch estratti dalle mammografie originali, seguendo la descrizione da ( http://arxiv.org/abs/1708.09427 ), in modo da inquadrare il compito da risolvere in un ambiente di classificazione delle immagini tradizionale.
Pagina iniziale : https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Codice sorgente :
tfds.image_classification.CuratedBreastImagingDDSMVersioni :
-
2.0.1: nuova API divisa ( https://tensorflow.org/datasets/splits ) -
3.0.0(impostazione predefinita): migliore campionamento del ritaglio ( https://github.com/tensorflow/datasets/pull/2502 )
-
Istruzioni per il download manuale : questo set di dati richiede il download manuale dei dati di origine in
download_config.manual_dir(il valore predefinito è~/tensorflow_datasets/downloads/manual/):
È possibile scaricare le immagini da https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Poiché per scaricare e leggere le immagini contenute nel set di dati sono necessari software e librerie speciali, TFDS presuppone che l'utente abbia scaricato i file DCIM originali e li abbia convertiti in PNG.
Per generare i file PNG è necessario utilizzare i seguenti comandi (o equivalenti) per garantire risultati riproducibili:
find $DATASET_DCIM_DIR -name '*.dcm' | \
xargs -n1 -P8 -I{} bash -c 'f={}; dcmj2pnm $f | convert - ${f/.dcm/.png}'
Le immagini risultanti dovrebbero essere inserite in manual_dir , come: <manual_dir>/Mass-Training_P_01981_RIGHT_MLO_1/1.3.6.../000000.png .
Memorizzazione nella cache automatica ( documentazione ): No
Chiavi supervisionate (vedi il documento
as_supervised):NoneCitazione :
@misc{CBIS_DDSM_Citation,
doi = {10.7937/k9/tcia.2016.7o02s9cy},
url = {https://wiki.cancerimagingarchive.net/x/lZNXAQ},
author = {Sawyer-Lee, Rebecca and Gimenez, Francisco and Hoogi, Assaf and Rubin, Daniel},
title = {Curated Breast Imaging Subset of DDSM},
publisher = {The Cancer Imaging Archive},
year = {2016},
}
@article{TCIA_Citation,
author = {
K. Clark and B. Vendt and K. Smith and J. Freymann and J. Kirby and
P. Koppel and S. Moore and S. Phillips and D. Maffitt and M. Pringle and
L. Tarbox and F. Prior
},
title = { {The Cancer Imaging Archive (TCIA): Maintaining and Operating a
Public Information Repository} },
journal = {Journal of Digital Imaging},
volume = {26},
month = {December},
year = {2013},
pages = {1045-1057},
}
@article{DBLP:journals/corr/abs-1708-09427,
author = {Li Shen},
title = {End-to-end Training for Whole Image Breast Cancer Diagnosis using
An All Convolutional Design},
journal = {CoRR},
volume = {abs/1708.09427},
year = {2017},
url = {http://arxiv.org/abs/1708.09427},
archivePrefix = {arXiv},
eprint = {1708.09427},
timestamp = {Mon, 13 Aug 2018 16:48:35 +0200},
biburl = {https://dblp.org/rec/bib/journals/corr/abs-1708-09427},
bibsource = {dblp computer science bibliography, https://dblp.org}
}
curated_breast_imaging_ddsm/patches (configurazione predefinita)
Descrizione della configurazione : patch contenenti sia casi di calcificazione che di massa, oltre a percorsi senza anomalie. Progettato come un tradizionale compito di classificazione in 5 classi.
Dimensione download :
2.01 MiBDimensione del set di dati :
801.46 MiBDivide :
| Diviso | Esempi |
|---|---|
'test' | 9.770 |
'train' | 49.780 |
'validation' | 5.580 |
- Struttura delle caratteristiche :
FeaturesDict({
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'label': ClassLabel(shape=(), dtype=int64, num_classes=5),
})
- Documentazione delle funzionalità :
| Caratteristica | Classe | Forma | Tipo D | Descrizione |
|---|---|---|---|---|
| CaratteristicheDict | ||||
| id | Testo | corda | ||
| Immagine | Immagine | (Nessuno, Nessuno, 1) | uint8 | |
| etichetta | ClassLabel | int64 |
- Figura ( tfds.show_examples ):
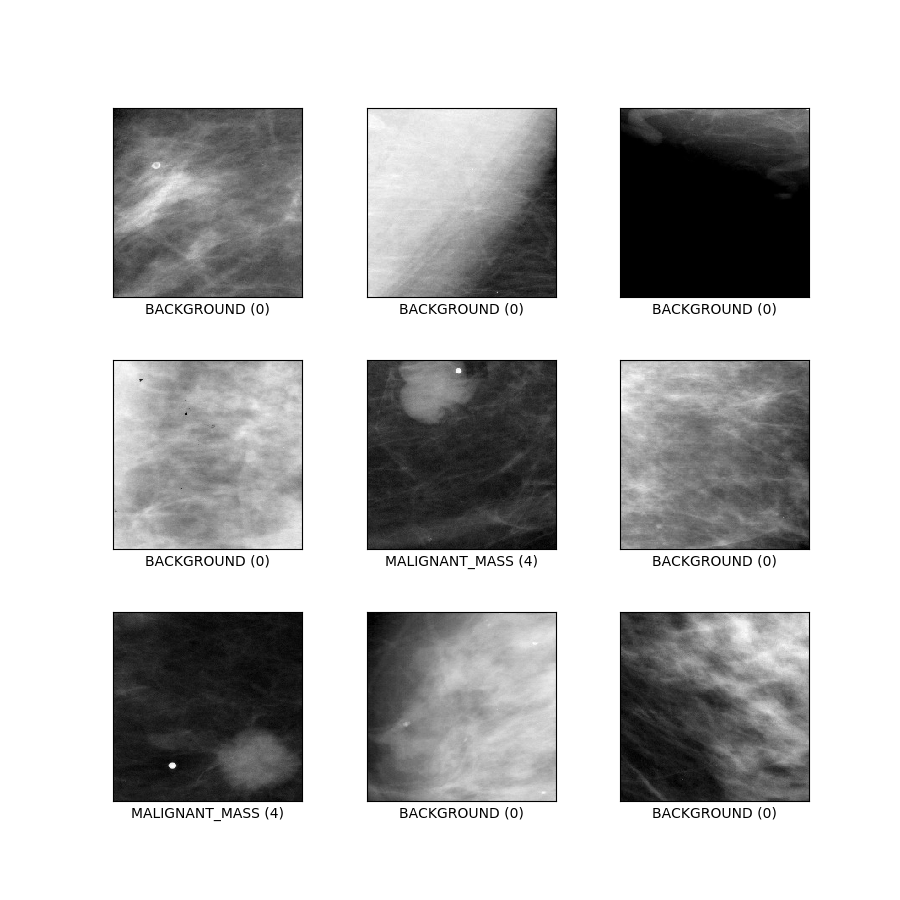
- Esempi ( tfds.as_dataframe ):
curated_breast_imaging_ddsm/original-calc
Descrizione configurazione : immagini originali dei casi di calcificazione compresse in PNG senza perdita di dati.
Dimensione download :
1.06 MiBDimensione del set di dati :
4.42 GiBDivide :
| Diviso | Esempi |
|---|---|
'test' | 284 |
'train' | 1.227 |
- Struttura delle caratteristiche :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'calc_distribution': ClassLabel(shape=(), dtype=int64, num_classes=10),
'calc_type': ClassLabel(shape=(), dtype=int64, num_classes=48),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentazione delle funzionalità :
| Caratteristica | Classe | Forma | Tipo D | Descrizione |
|---|---|---|---|---|
| CaratteristicheDict | ||||
| anomalie | Sequenza | |||
| anomalie/valutazione | ClassLabel | int64 | ||
| anomalie/calc_distribution | ClassLabel | int64 | ||
| anomalie/calc_type | ClassLabel | int64 | ||
| anomalie/id | Tensore | int32 | ||
| anomalie/maschera | Immagine | (Nessuno, Nessuno, 1) | uint8 | |
| anomalie/patologie | ClassLabel | int64 | ||
| anomalie/sottigliezza | ClassLabel | int64 | ||
| seno | ClassLabel | int64 | ||
| id | Testo | corda | ||
| Immagine | Immagine | (Nessuno, Nessuno, 1) | uint8 | |
| paziente | Testo | corda | ||
| visualizzazione | ClassLabel | int64 |
- Figura ( tfds.show_examples ):
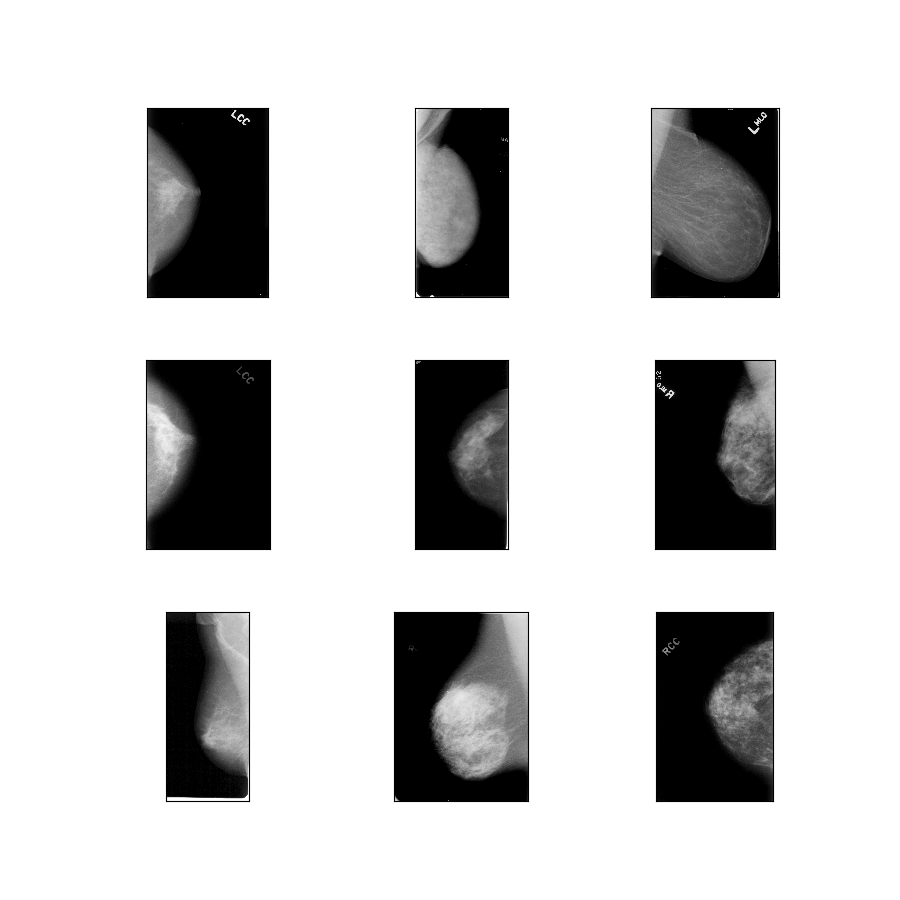
- Esempi ( tfds.as_dataframe ):
curated_breast_imaging_ddsm/original-mass
Descrizione configurazione : immagini originali dei casi di massa compressi in PNG senza perdita di dati.
Dimensione del download :
966.57 KiBDimensione del set di dati :
4.80 GiBDivide :
| Diviso | Esempi |
|---|---|
'test' | 348 |
'train' | 1.166 |
- Struttura delle caratteristiche :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'mass_margins': ClassLabel(shape=(), dtype=int64, num_classes=20),
'mass_shape': ClassLabel(shape=(), dtype=int64, num_classes=21),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentazione delle funzionalità :
| Caratteristica | Classe | Forma | Tipo D | Descrizione |
|---|---|---|---|---|
| CaratteristicheDict | ||||
| anomalie | Sequenza | |||
| anomalie/valutazione | ClassLabel | int64 | ||
| anomalie/id | Tensore | int32 | ||
| anomalie/maschera | Immagine | (Nessuno, Nessuno, 1) | uint8 | |
| anomalie/margini_di_massa | ClassLabel | int64 | ||
| anomalie/forma_massa | ClassLabel | int64 | ||
| anomalie/patologie | ClassLabel | int64 | ||
| anomalie/sottigliezza | ClassLabel | int64 | ||
| seno | ClassLabel | int64 | ||
| id | Testo | corda | ||
| Immagine | Immagine | (Nessuno, Nessuno, 1) | uint8 | |
| paziente | Testo | corda | ||
| visualizzazione | ClassLabel | int64 |
- Figura ( tfds.show_examples ):
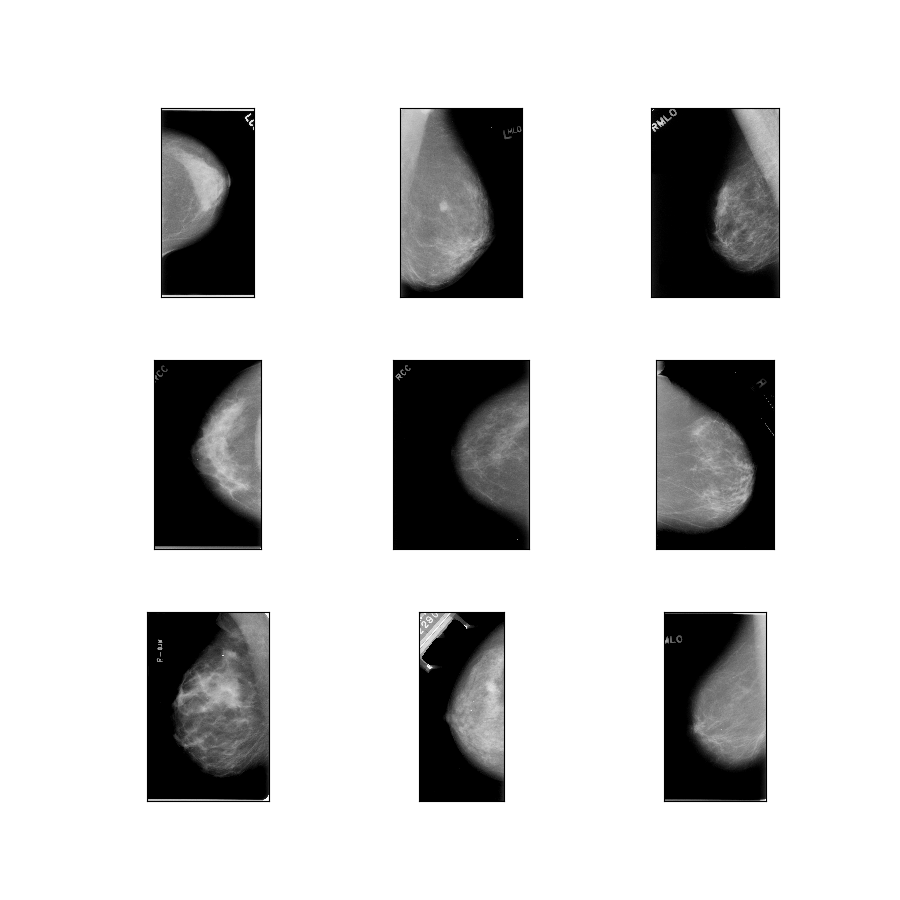
- Esempi ( tfds.as_dataframe ):

