- Description :
Le CBIS-DDSM (Curated Breast Imaging Subset of DDSM) est une version mise à jour et standardisée de la base de données numérique pour la mammographie de dépistage (DDSM). Le DDSM est une base de données de 2 620 études de mammographie sur film numérisé. Il contient des cas normaux, bénins et malins avec des informations pathologiques vérifiées.
La configuration par défaut est composée de correctifs extraits des mammographies originales, suivant la description de ( http://arxiv.org/abs/1708.09427 ), afin d'encadrer la tâche à résoudre dans un cadre de classification d'images traditionnel.
Page d'accueil : https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Code source :
tfds.image_classification.CuratedBreastImagingDDSMVersions :
-
2.0.1: Nouvelle API fractionnée ( https://tensorflow.org/datasets/splits ) -
3.0.0(par défaut) : meilleur échantillonnage de recadrage ( https://github.com/tensorflow/datasets/pull/2502 )
-
Instructions de téléchargement manuel : cet ensemble de données nécessite que vous téléchargiez les données sources manuellement dans
download_config.manual_dir(par défaut~/tensorflow_datasets/downloads/manual/) :
Vous pouvez télécharger les images depuis https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Étant donné que des logiciels et des bibliothèques spéciaux sont nécessaires pour télécharger et lire les images contenues dans l'ensemble de données, TFDS suppose que l'utilisateur a téléchargé les fichiers DCIM originaux et les a convertis en PNG.
Les commandes suivantes (ou équivalentes) doivent être utilisées pour générer les fichiers PNG, afin de garantir des résultats reproductibles :
find $DATASET_DCIM_DIR -name '*.dcm' | \
xargs -n1 -P8 -I{} bash -c 'f={}; dcmj2pnm $f | convert - ${f/.dcm/.png}'
Les images résultantes doivent être placées dans manual_dir , comme : <manual_dir>/Mass-Training_P_01981_RIGHT_MLO_1/1.3.6.../000000.png .
Mise en cache automatique ( documentation ) : Non
Clés supervisées (Voir doc
as_supervised) :NoneCitation :
@misc{CBIS_DDSM_Citation,
doi = {10.7937/k9/tcia.2016.7o02s9cy},
url = {https://wiki.cancerimagingarchive.net/x/lZNXAQ},
author = {Sawyer-Lee, Rebecca and Gimenez, Francisco and Hoogi, Assaf and Rubin, Daniel},
title = {Curated Breast Imaging Subset of DDSM},
publisher = {The Cancer Imaging Archive},
year = {2016},
}
@article{TCIA_Citation,
author = {
K. Clark and B. Vendt and K. Smith and J. Freymann and J. Kirby and
P. Koppel and S. Moore and S. Phillips and D. Maffitt and M. Pringle and
L. Tarbox and F. Prior
},
title = { {The Cancer Imaging Archive (TCIA): Maintaining and Operating a
Public Information Repository} },
journal = {Journal of Digital Imaging},
volume = {26},
month = {December},
year = {2013},
pages = {1045-1057},
}
@article{DBLP:journals/corr/abs-1708-09427,
author = {Li Shen},
title = {End-to-end Training for Whole Image Breast Cancer Diagnosis using
An All Convolutional Design},
journal = {CoRR},
volume = {abs/1708.09427},
year = {2017},
url = {http://arxiv.org/abs/1708.09427},
archivePrefix = {arXiv},
eprint = {1708.09427},
timestamp = {Mon, 13 Aug 2018 16:48:35 +0200},
biburl = {https://dblp.org/rec/bib/journals/corr/abs-1708-09427},
bibsource = {dblp computer science bibliography, https://dblp.org}
}
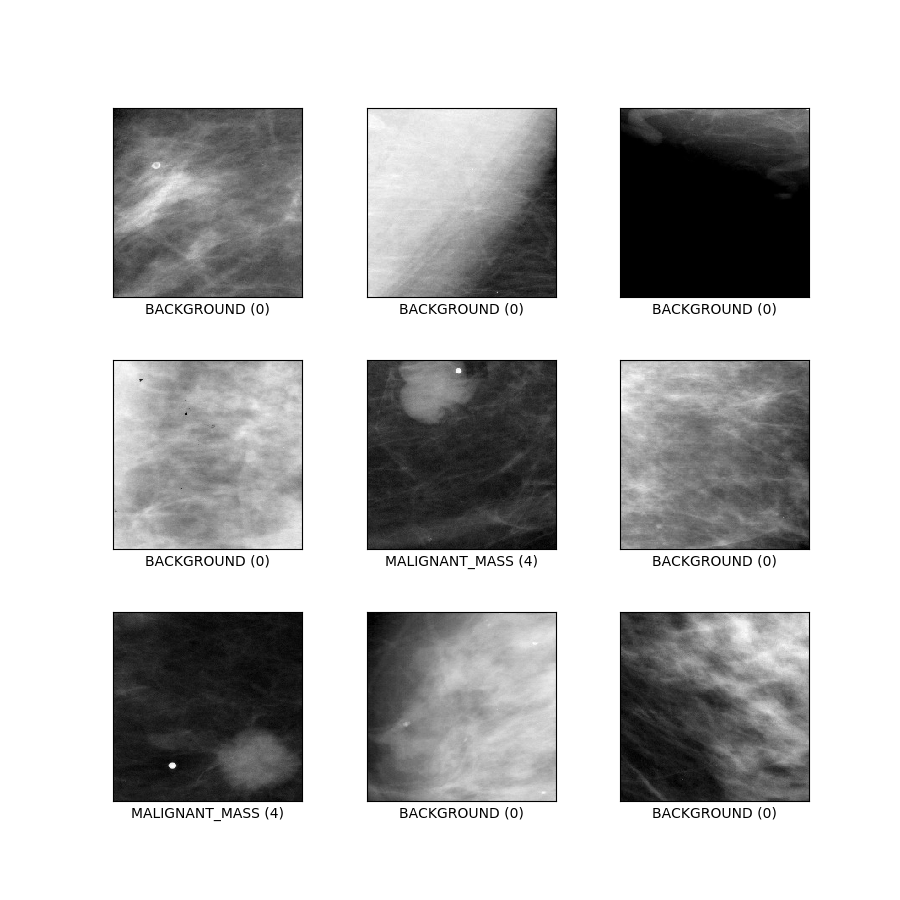
curated_breast_imaging_ddsm/patches (configuration par défaut)
Description de la configuration : Patchs contenant à la fois des cas de calcification et de masse, ainsi que des chemins sans anomalies. Conçu comme une tâche de classification traditionnelle en 5 classes.
Taille du téléchargement :
2.01 MiBTaille de l'ensemble de données :
801.46 MiBDivisions :
| Diviser | Exemples |
|---|---|
'test' | 9 770 |
'train' | 49 780 |
'validation' | 5 580 |
- Structure des fonctionnalités :
FeaturesDict({
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'label': ClassLabel(shape=(), dtype=int64, num_classes=5),
})
- Documentation des fonctionnalités :
| Fonctionnalité | Classe | Forme | Type D | Description |
|---|---|---|---|---|
| FonctionnalitésDict | ||||
| identifiant | Texte | chaîne | ||
| image | Image | (Aucun, Aucun, 1) | uint8 | |
| étiquette | Étiquette de classe | int64 |
- Figure ( tfds.show_examples ) :
- Exemples ( tfds.as_dataframe ) :
curated_breast_imaging_ddsm/original-calc
Description de la configuration : Images originales des cas de calcification compressées en PNG sans perte.
Taille du téléchargement :
1.06 MiBTaille de l'ensemble de données :
4.42 GiBDivisions :
| Diviser | Exemples |
|---|---|
'test' | 284 |
'train' | 1 227 |
- Structure des fonctionnalités :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'calc_distribution': ClassLabel(shape=(), dtype=int64, num_classes=10),
'calc_type': ClassLabel(shape=(), dtype=int64, num_classes=48),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentation des fonctionnalités :
| Fonctionnalité | Classe | Forme | Type D | Description |
|---|---|---|---|---|
| FonctionnalitésDict | ||||
| anomalies | Séquence | |||
| anomalies/évaluation | Étiquette de classe | int64 | ||
| anomalies/calc_distribution | Étiquette de classe | int64 | ||
| anomalies/calc_type | Étiquette de classe | int64 | ||
| anomalies/identifiant | Tenseur | int32 | ||
| anomalies/masque | Image | (Aucun, Aucun, 1) | uint8 | |
| anomalies/pathologie | Étiquette de classe | int64 | ||
| anomalies/subtilité | Étiquette de classe | int64 | ||
| sein | Étiquette de classe | int64 | ||
| identifiant | Texte | chaîne | ||
| image | Image | (Aucun, Aucun, 1) | uint8 | |
| patient | Texte | chaîne | ||
| voir | Étiquette de classe | int64 |
- Figure ( tfds.show_examples ) :
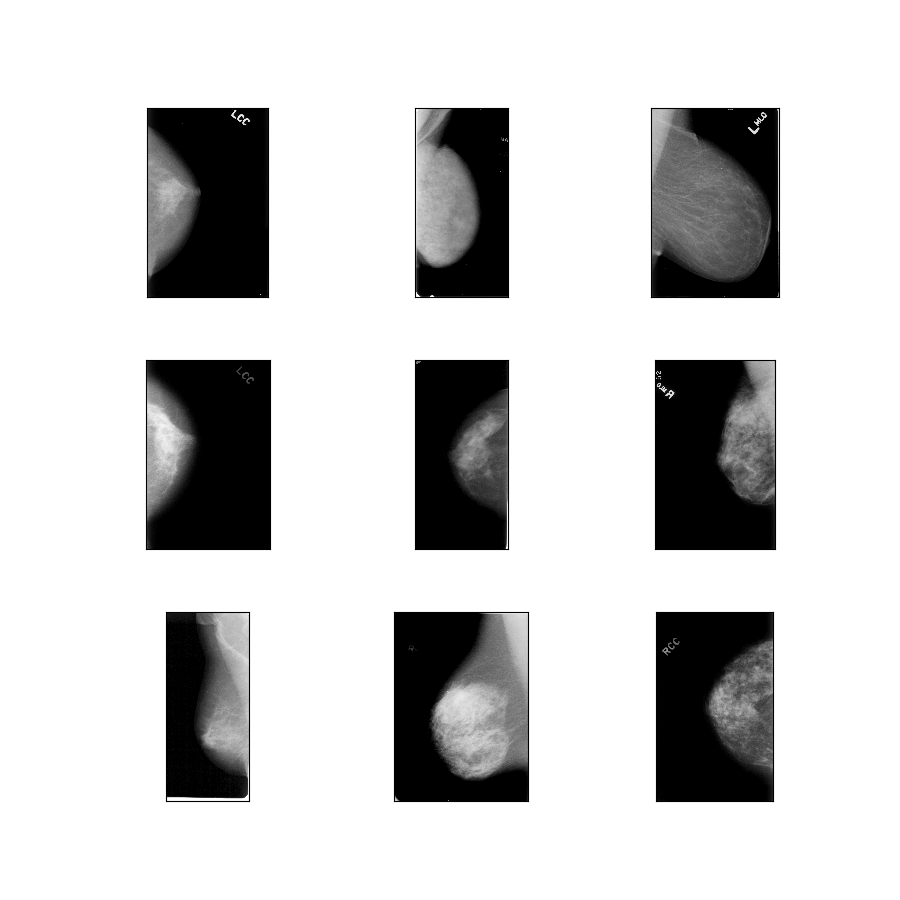
- Exemples ( tfds.as_dataframe ) :
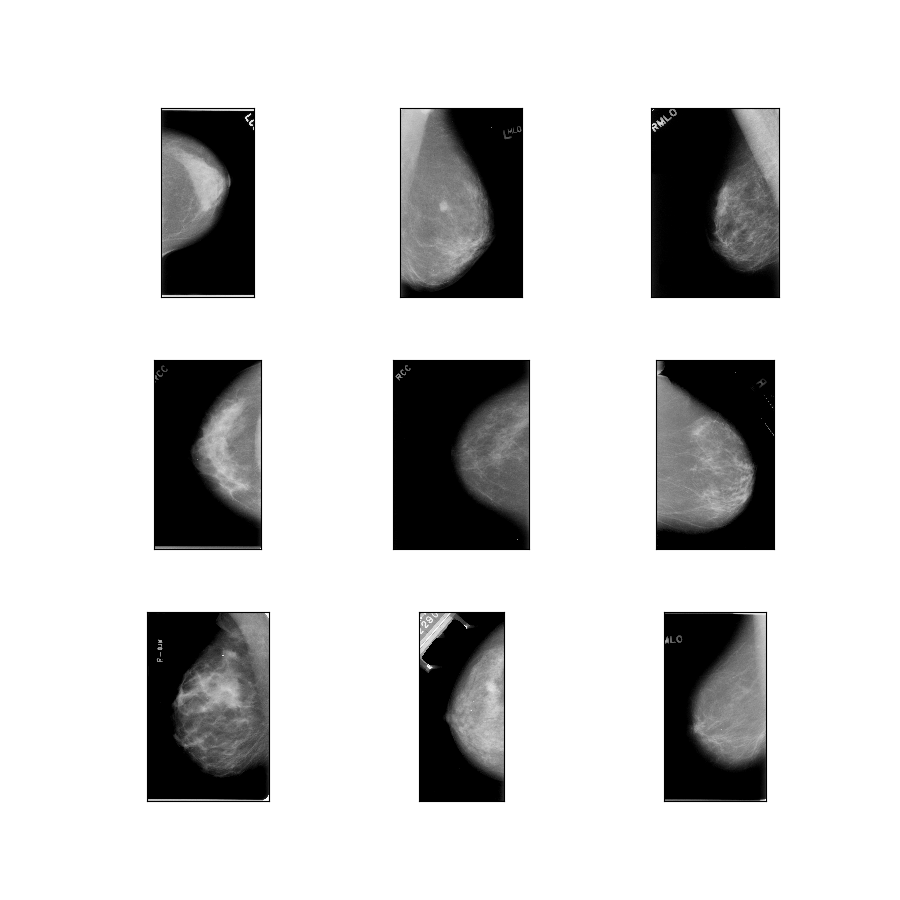
curated_breast_imaging_ddsm/original-mass
Description de la configuration : Images originales des cas de masse compressées en PNG sans perte.
Taille du téléchargement :
966.57 KiBTaille du jeu de données :
4.80 GiBDivisions :
| Diviser | Exemples |
|---|---|
'test' | 348 |
'train' | 1 166 |
- Structure des fonctionnalités :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'mass_margins': ClassLabel(shape=(), dtype=int64, num_classes=20),
'mass_shape': ClassLabel(shape=(), dtype=int64, num_classes=21),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentation des fonctionnalités :
| Fonctionnalité | Classe | Forme | Type D | Description |
|---|---|---|---|---|
| FonctionnalitésDict | ||||
| anomalies | Séquence | |||
| anomalies/évaluation | Étiquette de classe | int64 | ||
| anomalies/identifiant | Tenseur | int32 | ||
| anomalies/masque | Image | (Aucun, Aucun, 1) | uint8 | |
| anomalies/marges_de masse | Étiquette de classe | int64 | ||
| anomalies/mass_shape | Étiquette de classe | int64 | ||
| anomalies/pathologie | Étiquette de classe | int64 | ||
| anomalies/subtilité | Étiquette de classe | int64 | ||
| sein | Étiquette de classe | int64 | ||
| identifiant | Texte | chaîne | ||
| image | Image | (Aucun, Aucun, 1) | uint8 | |
| patient | Texte | chaîne | ||
| voir | Étiquette de classe | int64 |
- Figure ( tfds.show_examples ) :
- Exemples ( tfds.as_dataframe ) :

