- Opis :
CBIS-DDSM (Curated Breast Imaging Subset of DDSM) to zaktualizowana i ujednolicona wersja cyfrowej bazy danych mammografii przesiewowej (DDSM). DDSM to baza danych zawierająca 2620 badań mammograficznych na kliszy skanowanej. Zawiera przypadki normalne, łagodne i złośliwe ze zweryfikowanymi informacjami o patologii.
Domyślna konfiguracja składa się z fragmentów wyodrębnionych z oryginalnych mammogramów, zgodnie z opisem z ( http://arxiv.org/abs/1708.09427 ), aby określić zadanie do rozwiązania w ramach tradycyjnego ustawienia klasyfikacji obrazów.
Strona główna : https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Kod źródłowy :
tfds.image_classification.CuratedBreastImagingDDSMWersje :
-
2.0.1: Nowe podzielone API ( https://tensorflow.org/datasets/splits ) -
3.0.0(domyślnie): Lepsze próbkowanie przycinania ( https://github.com/tensorflow/datasets/pull/2502 )
-
Instrukcje ręcznego pobierania : ten zestaw danych wymaga ręcznego pobrania danych źródłowych do
download_config.manual_dir(domyślnie jest to~/tensorflow_datasets/downloads/manual/):
Obrazy możesz pobrać z https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Ponieważ do pobrania i odczytania obrazów zawartych w zbiorze danych potrzebne jest specjalne oprogramowanie i biblioteki, TFDS zakłada, że użytkownik pobrał oryginalne pliki DCIM i przekonwertował je do formatu PNG.
Aby wygenerować pliki PNG, należy użyć następujących poleceń (lub ich odpowiedników), aby zagwarantować powtarzalność wyników:
find $DATASET_DCIM_DIR -name '*.dcm' | \
xargs -n1 -P8 -I{} bash -c 'f={}; dcmj2pnm $f | convert - ${f/.dcm/.png}'
Wynikowe obrazy należy umieścić w manual_dir , np.: <manual_dir>/Mass-Training_P_01981_RIGHT_MLO_1/1.3.6.../000000.png .
Automatyczne buforowanie ( dokumentacja ): Nie
Klucze nadzorowane (zobacz dokument
as_supervised):NoneCytat :
@misc{CBIS_DDSM_Citation,
doi = {10.7937/k9/tcia.2016.7o02s9cy},
url = {https://wiki.cancerimagingarchive.net/x/lZNXAQ},
author = {Sawyer-Lee, Rebecca and Gimenez, Francisco and Hoogi, Assaf and Rubin, Daniel},
title = {Curated Breast Imaging Subset of DDSM},
publisher = {The Cancer Imaging Archive},
year = {2016},
}
@article{TCIA_Citation,
author = {
K. Clark and B. Vendt and K. Smith and J. Freymann and J. Kirby and
P. Koppel and S. Moore and S. Phillips and D. Maffitt and M. Pringle and
L. Tarbox and F. Prior
},
title = { {The Cancer Imaging Archive (TCIA): Maintaining and Operating a
Public Information Repository} },
journal = {Journal of Digital Imaging},
volume = {26},
month = {December},
year = {2013},
pages = {1045-1057},
}
@article{DBLP:journals/corr/abs-1708-09427,
author = {Li Shen},
title = {End-to-end Training for Whole Image Breast Cancer Diagnosis using
An All Convolutional Design},
journal = {CoRR},
volume = {abs/1708.09427},
year = {2017},
url = {http://arxiv.org/abs/1708.09427},
archivePrefix = {arXiv},
eprint = {1708.09427},
timestamp = {Mon, 13 Aug 2018 16:48:35 +0200},
biburl = {https://dblp.org/rec/bib/journals/corr/abs-1708-09427},
bibsource = {dblp computer science bibliography, https://dblp.org}
}
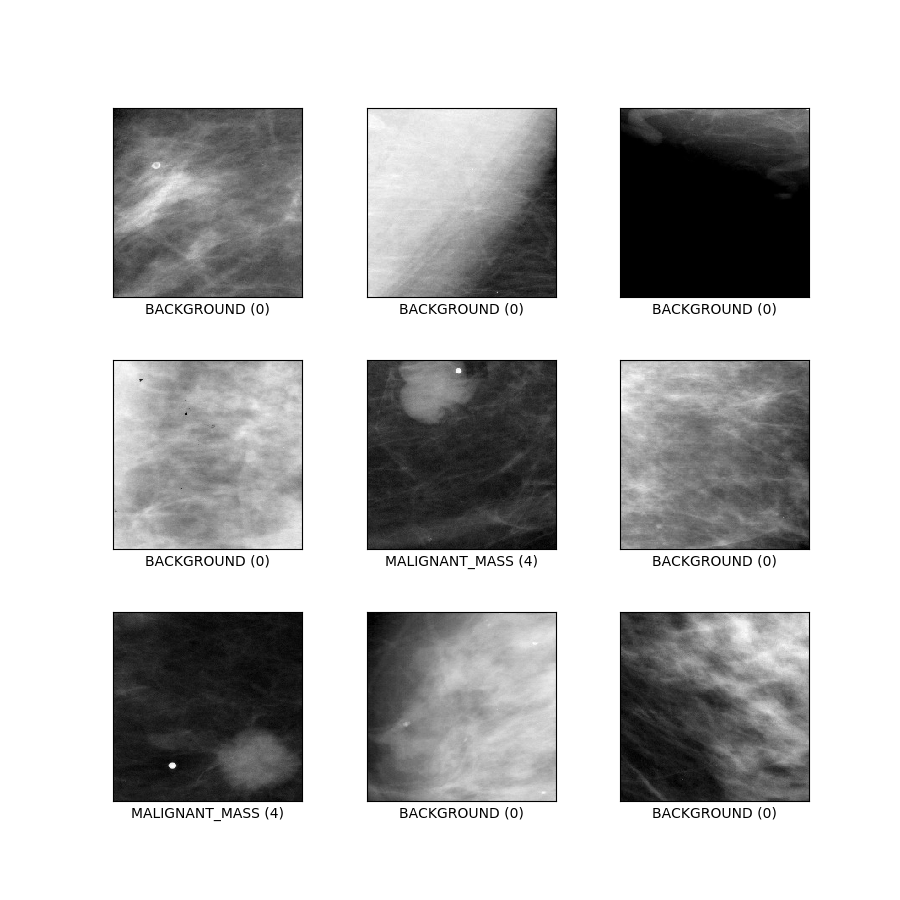
curated_breast_imaging_ddsm/patches (konfiguracja domyślna)
Opis konfiguracji : Poprawki zawierające przypadki zwapnienia i masy, a także ścieżki bez nieprawidłowości. Zaprojektowane jako tradycyjne zadanie klasyfikacji 5-klasowej.
Rozmiar pliku do pobrania :
2.01 MiBRozmiar zbioru danych :
801.46 MiBPodziały :
| Podział | Przykłady |
|---|---|
'test' | 9770 |
'train' | 49780 |
'validation' | 5580 |
- Struktura funkcji :
FeaturesDict({
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'label': ClassLabel(shape=(), dtype=int64, num_classes=5),
})
- Dokumentacja funkcji :
| Funkcja | Klasa | Kształt | Typ D | Opis |
|---|---|---|---|---|
| FunkcjeDykt | ||||
| ID | Tekst | strunowy | ||
| obraz | Obraz | (Brak, Brak, 1) | uint8 | |
| etykieta | Etykieta klasy | int64 |
- Rysunek ( tfds.show_examples ):
- Przykłady ( tfds.as_dataframe ):
curated_breast_imaging_ddsm/original-calc
Opis konfiguracji : Oryginalne obrazy przypadków zwapnień skompresowane w bezstratnym formacie PNG.
Rozmiar pliku do pobrania :
1.06 MiBRozmiar zbioru danych :
4.42 GiBPodziały :
| Podział | Przykłady |
|---|---|
'test' | 284 |
'train' | 1227 |
- Struktura funkcji :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'calc_distribution': ClassLabel(shape=(), dtype=int64, num_classes=10),
'calc_type': ClassLabel(shape=(), dtype=int64, num_classes=48),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Dokumentacja funkcji :
| Funkcja | Klasa | Kształt | Typ D | Opis |
|---|---|---|---|---|
| FunkcjeDykt | ||||
| nieprawidłowości | Sekwencja | |||
| nieprawidłowości/ocena | Etykieta klasy | int64 | ||
| nieprawidłowości/calc_distribution | Etykieta klasy | int64 | ||
| nieprawidłowości/typ_calc | Etykieta klasy | int64 | ||
| nieprawidłowości/id | Napinacz | int32 | ||
| nieprawidłowości/maska | Obraz | (Brak, Brak, 1) | uint8 | |
| nieprawidłowości/patologia | Etykieta klasy | int64 | ||
| nieprawidłowości/subtelność | Etykieta klasy | int64 | ||
| pierś | Etykieta klasy | int64 | ||
| ID | Tekst | strunowy | ||
| obraz | Obraz | (Brak, Brak, 1) | uint8 | |
| pacjent | Tekst | strunowy | ||
| pogląd | Etykieta klasy | int64 |
- Rysunek ( tfds.show_examples ):
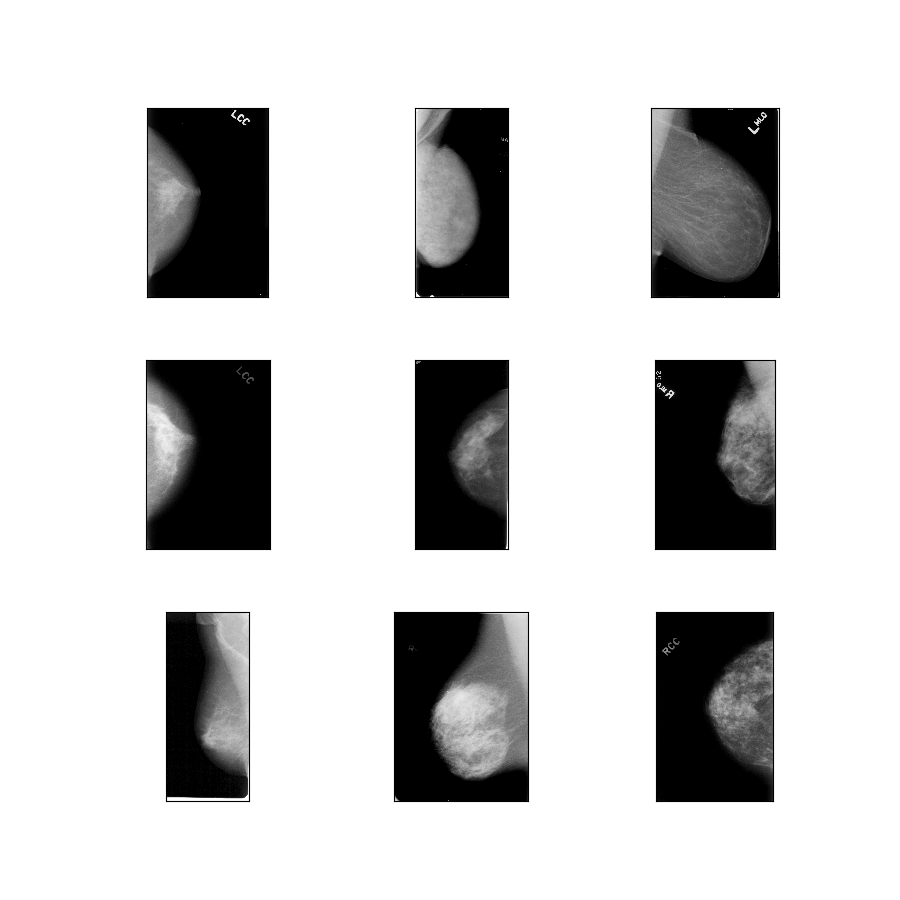
- Przykłady ( tfds.as_dataframe ):
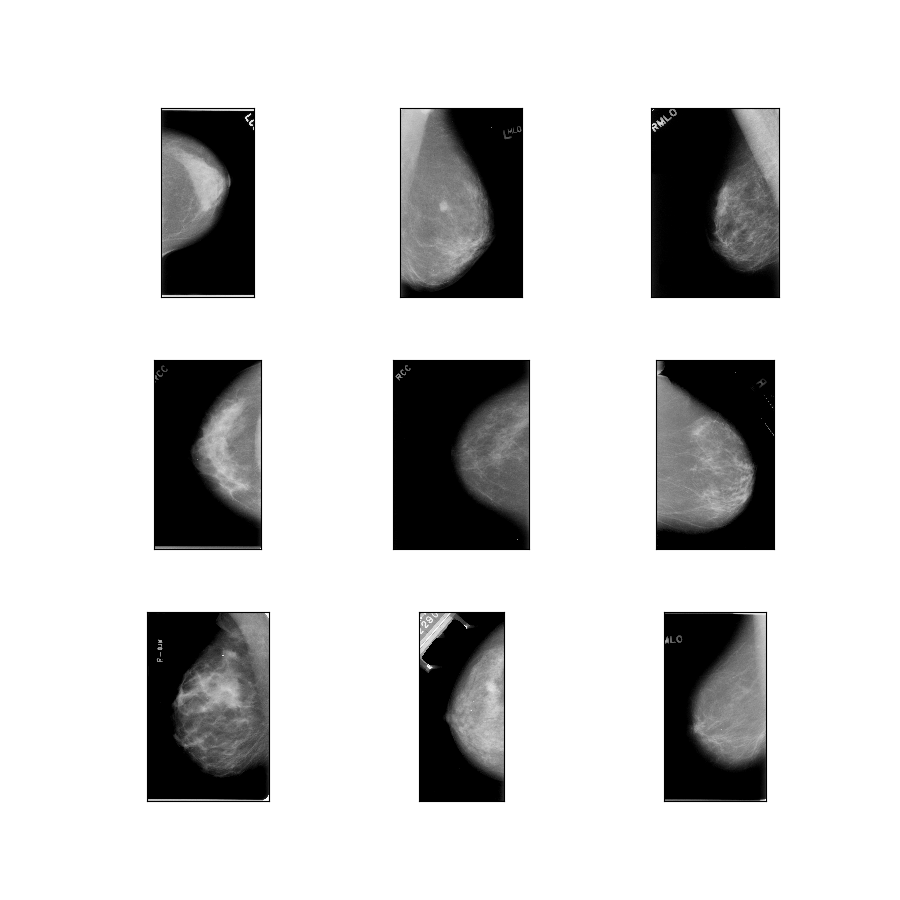
curated_breast_imaging_ddsm/original-mass
Opis konfiguracji : Oryginalne obrazy masowych przypadków skompresowane w bezstratnym formacie PNG.
Rozmiar pliku do pobrania :
966.57 KiBRozmiar zbioru danych :
4.80 GiBPodziały :
| Podział | Przykłady |
|---|---|
'test' | 348 |
'train' | 1166 |
- Struktura funkcji :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'mass_margins': ClassLabel(shape=(), dtype=int64, num_classes=20),
'mass_shape': ClassLabel(shape=(), dtype=int64, num_classes=21),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Dokumentacja funkcji :
| Funkcja | Klasa | Kształt | Typ D | Opis |
|---|---|---|---|---|
| FunkcjeDykt | ||||
| nieprawidłowości | Sekwencja | |||
| nieprawidłowości/ocena | Etykieta klasy | int64 | ||
| nieprawidłowości/id | Napinacz | int32 | ||
| nieprawidłowości/maska | Obraz | (Brak, Brak, 1) | uint8 | |
| nieprawidłowości/marginesy_masy | Etykieta klasy | int64 | ||
| nieprawidłowości/kształt_masy | Etykieta klasy | int64 | ||
| nieprawidłowości/patologia | Etykieta klasy | int64 | ||
| nieprawidłowości/subtelność | Etykieta klasy | int64 | ||
| pierś | Etykieta klasy | int64 | ||
| ID | Tekst | strunowy | ||
| obraz | Obraz | (Brak, Brak, 1) | uint8 | |
| pacjent | Tekst | strunowy | ||
| pogląd | Etykieta klasy | int64 |
- Rysunek ( tfds.show_examples ):
- Przykłady ( tfds.as_dataframe ):

