- Description :
Classification des textures en histologie du cancer colorectal. Chaque exemple est une image RVB 150 x 150 x 3 de l'une des 8 classes.
Page d'accueil : https://zenodo.org/record/53169#.XGZemKwzbmG
Code source :
tfds.image_classification.ColorectalHistologyVersions :
-
2.0.0(par défaut) : nouvelle API fractionnée ( https://tensorflow.org/datasets/splits )
-
Taille du téléchargement :
246.14 MiBTaille de l'ensemble de données :
179.23 MiBMise en cache automatique ( documentation ) : uniquement lorsque
shuffle_files=False(train)Divisions :
| Diviser | Exemples |
|---|---|
'train' | 5 000 |
- Structure des fonctionnalités :
FeaturesDict({
'filename': Text(shape=(), dtype=string),
'image': Image(shape=(150, 150, 3), dtype=uint8),
'label': ClassLabel(shape=(), dtype=int64, num_classes=8),
})
- Documentation des fonctionnalités :
| Fonctionnalité | Classe | Forme | Type D | Description |
|---|---|---|---|---|
| FonctionnalitésDict | ||||
| nom de fichier | Texte | chaîne | ||
| image | Image | (150, 150, 3) | uint8 | |
| étiquette | Étiquette de classe | int64 | Huit classes : 0 : « épithélium tumoral », 1 : « stroma simple », 2 : « stroma complexe » (stroma contenant des cellules tumorales uniques et/ou des cellules immunitaires uniques), 3 : « conglomérats de cellules immunitaires », 4 : « débris et mucus', 5 : 'glandes muqueuses', 6 : 'tissu adipeux' et 7 : 'fond'. |
Clés supervisées (Voir doc
as_supervised) :('image', 'label')Figure ( tfds.show_examples ) :
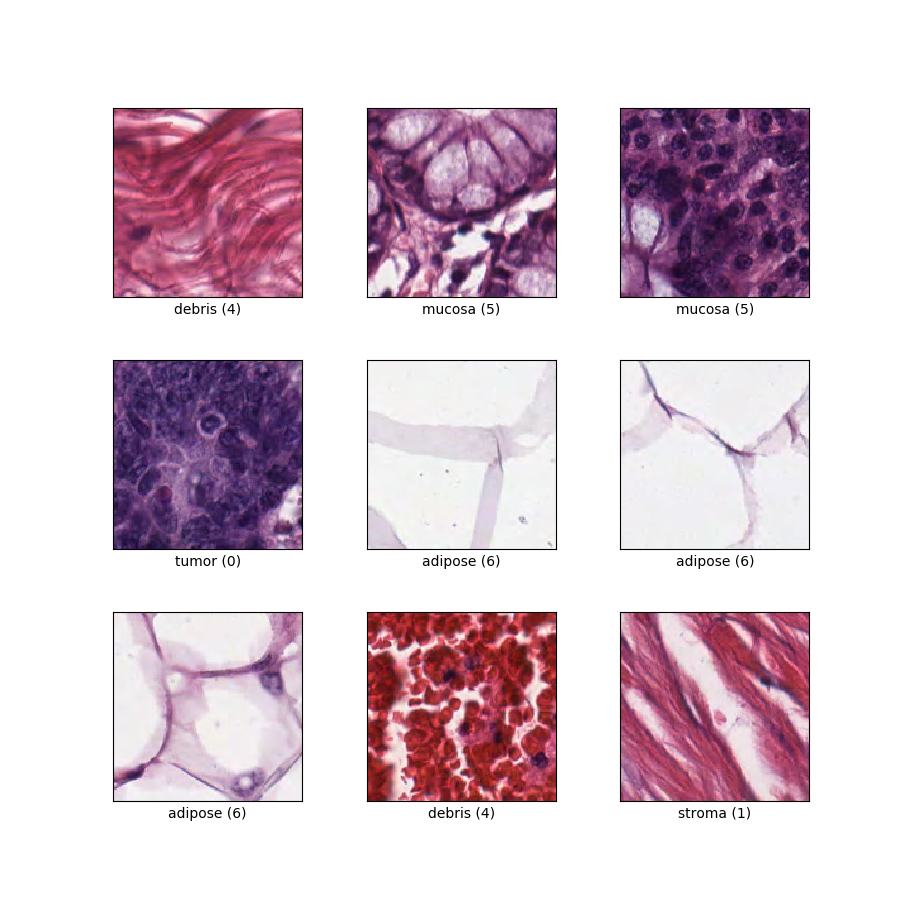
- Exemples ( tfds.as_dataframe ) :
- Citation :
@article{kather2016multi,
title={Multi-class texture analysis in colorectal cancer histology},
author={Kather, Jakob Nikolas and Weis, Cleo-Aron and Bianconi, Francesco and Melchers, Susanne M and Schad, Lothar R and Gaiser, Timo and Marx, Alexander and Z{"o}llner, Frank Gerrit},
journal={Scientific reports},
volume={6},
pages={27988},
year={2016},
publisher={Nature Publishing Group}
}

